朴素贝叶斯方法(Naive Bayes Method)
2015-07-24 11:23
981 查看
朴素贝叶斯是一种很简单的分类方法,之所以称之为朴素,是因为它有着非常强的前提条件-其所有特征都是相互独立的,是一种典型的生成学习算法。所谓生成学习算法,是指由训练数据学习联合概率分布P(X,Y),然后求得后验概率P(X|Y)。具体来说,利用训练数据学习P(X|Y)和p(Y)的估计,得到联合概率分布:

概率估计可以是极大似然估计,或者贝叶斯估计。
假设输入 X 为n维的向量集合,输出 Y 为类别,X 和 Y 都是随机变量。P(X,Y)是X和Y的联合概率分布,训练数据集为:

首先,我们要明确我们求解的目标是:

,即给定某个输入X,我们要判断其所属类别Ck。由概率论知识,我们有:
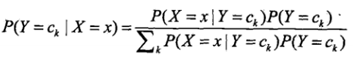
其中,

代入公式得:
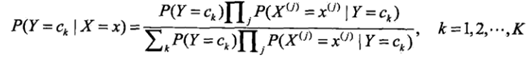
这是朴素贝叶斯分类的基本公式。于是,朴素贝叶斯分类器可以表示为
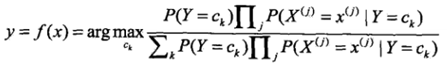
由于,分母对所有的Ck都是相同的,所以

那么如果给定一个输入 X,我们只需要找到一个类别Ck,使得

最大。那么Ck,就是 X 的最佳类别了。
下面我们来讲讲朴素贝叶斯法的参数估计,为什么要估计朴素贝叶斯的参数呢,这些参数是什么?首先,我们要明确。现实中,给定我们一批数据,我们就知道其分布,但是具体的数据分布的概率我们是不知道的。也就是说先验概率和条件概率我们是不知道的,这就需要我们来利用其数据的分布估计其先验概率和条件概率了。统计学习中最常用的参数估计就是极大似然估计了,这里我们也可以用贝叶斯估计,其实就是在极大似然估计基础上添加了拉普拉斯平滑(Laplace smoothing)。
由于极大似然估计之前已经讲到过,这里公式我也没有具体来推,所以先验概率和条件概率直接给出来。
先验概率P(Y = Ck)和条件概率的极大似然估计如下:
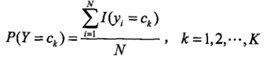

这样,给定具体的数据,我们就可以估计其先验概率和条件概率,进而计算出后验概率得到所属类别。
同样,贝叶斯估计和极大似然估计差不多,贝叶斯估计只是在极大似然估计上添加了一个拉普拉斯平滑。具体如下:
条件概率的贝叶斯估计如下:
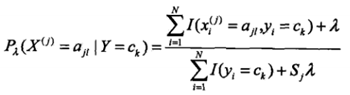
先验概率的贝叶斯估计如下:
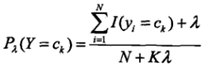
下面来给出一个简单的朴素贝叶斯实现代码,代码比较容易理解。只是课本上给出的特征是离散的,而code里面的特征是连续的。原理上其实是一样一样的~
如果想要实验数据的话,请在博客下面评论区域注明,我看到了会第一时间上传。

概率估计可以是极大似然估计,或者贝叶斯估计。
假设输入 X 为n维的向量集合,输出 Y 为类别,X 和 Y 都是随机变量。P(X,Y)是X和Y的联合概率分布,训练数据集为:

首先,我们要明确我们求解的目标是:

,即给定某个输入X,我们要判断其所属类别Ck。由概率论知识,我们有:
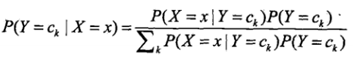
其中,

代入公式得:
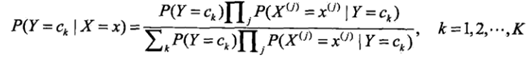
这是朴素贝叶斯分类的基本公式。于是,朴素贝叶斯分类器可以表示为
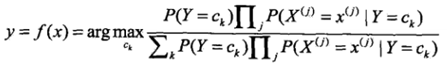
由于,分母对所有的Ck都是相同的,所以

那么如果给定一个输入 X,我们只需要找到一个类别Ck,使得

最大。那么Ck,就是 X 的最佳类别了。
下面我们来讲讲朴素贝叶斯法的参数估计,为什么要估计朴素贝叶斯的参数呢,这些参数是什么?首先,我们要明确。现实中,给定我们一批数据,我们就知道其分布,但是具体的数据分布的概率我们是不知道的。也就是说先验概率和条件概率我们是不知道的,这就需要我们来利用其数据的分布估计其先验概率和条件概率了。统计学习中最常用的参数估计就是极大似然估计了,这里我们也可以用贝叶斯估计,其实就是在极大似然估计基础上添加了拉普拉斯平滑(Laplace smoothing)。
由于极大似然估计之前已经讲到过,这里公式我也没有具体来推,所以先验概率和条件概率直接给出来。
先验概率P(Y = Ck)和条件概率的极大似然估计如下:
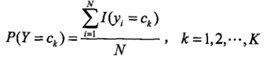

这样,给定具体的数据,我们就可以估计其先验概率和条件概率,进而计算出后验概率得到所属类别。
同样,贝叶斯估计和极大似然估计差不多,贝叶斯估计只是在极大似然估计上添加了一个拉普拉斯平滑。具体如下:
条件概率的贝叶斯估计如下:
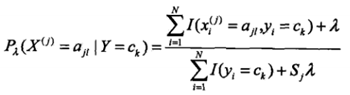
先验概率的贝叶斯估计如下:
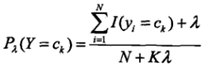
下面来给出一个简单的朴素贝叶斯实现代码,代码比较容易理解。只是课本上给出的特征是离散的,而code里面的特征是连续的。原理上其实是一样一样的~
% NAIVE BAYES CLASSIFIER
clear
tic
disp('--- start ---')distr='normal';
distr='kernel';
% read data
White_Wine = dataset('xlsfile', 'White_Wine.xlsx');X = double(White_Wine(:,1:11));
Y = double(White_Wine(:,12));
% Create a cvpartition object that defined the folds
c = cvpartition(Y,'holdout',.2);
% Create a training set
x = X(training(c,1),:);
y = Y(training(c,1));
% test set
u=X(test(c,1),:);
v=Y(test(c,1),:);
yu=unique(y);
nc=length(yu); % number of classes
ni=size(x,2); % independent variables
ns=length(v);% test set
% compute class probability
for i=1:nc
fy(i)=sum(double(y==yu(i)))/length(y);
end
switch distr
case 'normal'
% normal distribution
% parameters from training set
for i=1:nc
xi=x((y==yu(i)),:);
mu(i,:)=mean(xi,1);
sigma(i,:)=std(xi,1);
end
% probability for test set
for j=1:ns
fu=normcdf(ones(nc,1)*u(j,:),mu,sigma);
P(j,:)=fy.*prod(fu,2)';
end
case 'kernel'
% kernel distribution
% probability of test set estimated from training set
for i=1:nc
for k=1:ni
xi=x(y==yu(i),k);%the feature of dimension-k with respect to label yu(i)
ui=u(:,k);
fuStruct(i,k).f=ksdensity(xi,ui);
end
end
% re-structure
for i=1:ns
for j=1:nc
for k=1:ni
fu(j,k)=fuStruct(j,k).f(i);
end
end
P(i,:)=fy.*prod(fu,2)';
end
otherwise
disp('invalid distribution stated')return
end
% get predicted output for test set
[pv0,id]=max(P,[],2);
for i=1:length(id)
pv(i,1)=yu(id(i));
end
% compare predicted output with actual output from test data
confMat=myconfusionmat(v,pv);
disp('confusion matrix:')disp(confMat)
conf=sum(pv==v)/length(pv);
disp(['accuracy = ',num2str(conf*100),'%'])
toc
function confMat=myconfusionmat(v,pv)
yu=unique(v);
confMat=zeros(length(yu));
for i=1:length(yu)
for j=1:length(yu)
confMat(i,j)=sum(v==yu(i) & pv==yu(j));
end
end
如果想要实验数据的话,请在博客下面评论区域注明,我看到了会第一时间上传。
相关文章推荐
- mybatis 批量增加 Parameter '__frch_item_0' not found. Available parameters are [list]
- Algorithms—24.Swap Nodes in Pairs
- <学习笔记> public static void main(String[] args)小结
- linker command failed with exit code 1 错误小结
- hdu1151 air Raid(最小路径覆盖)
- [ 2015多校联合训练赛 hdu 5308 I Wanna Become A 24-Point Master 2015 Multi-University Training Contest 2 模拟题
- 10亿美金的教训——我居然错过Airbnb的天使轮
- Genymotion出现错误INSTALL_FAILED_CPU_ABI_INCOMPATIBLE解决办法
- AIX主机信任关系配置
- hdu 5308 I Wanna Become A 24-Point Master(2015 Multi-University Training Contest 2)
- sendmail笔记
- RAID在企业服务器中的应用(RAID几种级别)
- VMware vmdk错误: Failed to lock the file
- 16个网站 --- 免费的人工智能电子书
- Quartz 2.2.1学习笔记 (二) Jobs、JobDetail、JobDataMap
- maya之3d paint tool(3d绘制工具)
- HDU 5294 Tricks Device 2015 Multi-University Training Contest 1 07
- hdu 5308 I Wanna Become A 24-Point Master 2015 Multi-University Training Contest 2
- 2015 HUAS Summer Training#2~C
- hdu 3461 Code Lock(并查集)2010 ACM-ICPC Multi-University Training Contest(3)
