Pandas画图、统计、累计、csv文件的读取,缺失值的处理
2020-07-29 23:20
501 查看
Pandas画图:
api手册如下:


具体演示:
准备数据:
- data = pd.read_csv("./data/stock_day.csv")
- data = data.drop([“ma5”, “ma10”, “ma20”, “v_ma5”, “v_ma10”, “v_ma20”], axis=1)

绘制散点图:

- 通过上图我们发现上面这两个字段貌似没有什么内在的关系。

- 通过画图我们发现上面这两个字段貌似你在增长的时候我也在增长。

pandas的IO操作
- 我们的数据大部分存在于文件当中,所以pandas会支持复杂的IO操作,pandas的API支持众多的文件格式,如CSV、SQL、XLS、JSON、HDF5。

csv文件的读取于储存:

读取之前的股票数据:
- data = pd.read_csv("./data/stock_day.csv")

- data = data.drop([“ma5”, “ma10”, “ma20”, “v_ma5”, “v_ma10”, “v_ma20”], axis=1)


- 也可以在读取数据的时候指定读取的字段
- data = pd.read_csv("./data/stock_day.csv", usecols=[“open”, “high”, “close”])

也可以在读取文件的时候设置列索引:
- names=[“open”, “high”, “close”, “low”, “volume”, “price_change”, “p_change”, “ma5”, “ma10”, “ma20”, “v_ma5”, “v_ma10”, “v_ma20”, “turnover”]

将数据写入到csv文件中

- 选取前10行数据保存,便于观察数据:

- data[:10].to_csv("./data/test.csv", columns=[“open”])



读取、查看结果:
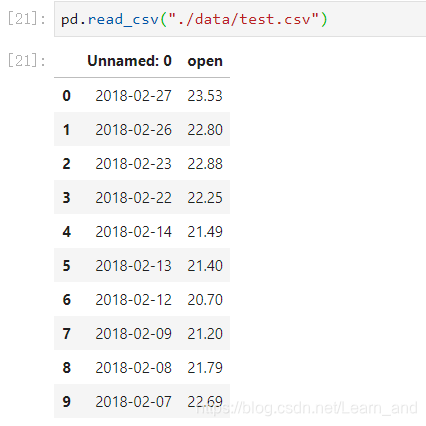
- 会发现将索引存入到文件当中,变成单独的一列数据。如果需要删除,可以指定index参数,删除原来的文件,重新保存一次。


指定追加方式:



- 又存进了一个列名,所以当以追加方式添加数据的时候,一定要去掉列名columns,指定header=False


hdf5文件的读取与存储:

文件的读取:

需要安装tables模块避免不能读取HDF5文件:










- 将读取到的day_high数据再写入到test.h5文件中:


- 注意:此时再来读取的时候必须要指定key了,因为现在这个h5文件中有两份数据。



json文件的读取与存储:

数据介绍:


读取数据:



写入数据:






- 默认是lines=False:


总结:

处理np.nan类型的缺失值:

如何处理nan:

缺失值处理案例:
- 电影数据文件获取:


- 判断缺失值是否存在(也就是判断是否存在nan类型的缺失值):


- 或是:


- 也可以:

- 说明:Revenue和Metascore这两个字段是有缺失值的。


缺失值处理方法1:删除含有缺失值的样本。

- 注意:dropna()方法不会修改源数据,需要接收返回值。

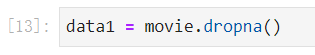

- 提示:添加inplace=True的话就是在原数据集上进行修改。
缺失值处理方法2:使用这一列的均值进行填充。

- 再来查看一下:

处理其他标记的缺失值:
- 不是缺失值nan,有默认标记的。

- 数据集介绍:威斯康星州乳腺癌数据库(1991年1月8日)
https://archive.ics.uci.edu/ml/machine-learning-databases/breast-cancer-wisconsin/



- 可以使用浏览器上的在线翻译功能进行翻译一下:



- path = “https://archive.ics.uci.edu/ml/machine-learning-databases/breast-cancer-wisconsin/breast-cancer-wisconsin.data”
- names = [“Sample code number”, “Clump Thickness”, “Uniformity of Cell Size”, “Uniformity of Cell Shape”, “Marginal Adhesion”, “Single Epithelial Cell Size”, “Bare Nuclei”, “Bland Chromatin”, “Normal Nucleoli”, “Mitoses”, “Class”]


处理思路分析:



具体演示:



总结:


相关文章推荐
- 利用pandas模块读取csv文件和excel表格,并用matplotlib画图
- pandas读取csv文件进行处理时报错: TypeError: invalid type comparison
- 使用pandas模块读取csv文件和excel表格,并用matplotlib画图的方法
- pandas读取csv文件进行处理时报错: TypeError: invalid type compariso
- pandas读取csv文件进行处理时报错: TypeError: invalid type comparison
- 读取csv文件并使用pandas.Series.apply进行处理时,对header=?的处理
- 操作CSV文件例子,并且加上文字处理:统计相同功能的问题单(报告画图用)
- pandas读取csv文件进行处理时报错: TypeError: invalid type comparison
- PHP按行读取、处理较大CSV文件的代码实例
- pandas 如何读取csv文件
- pandas.read_csv——分块读取大文件
- Python Pandas批量读取csv文件到dataframe的方法
- Pandas Python读取CSV文件中的某一列
- pandas读取csv文件报错
- 利用numpy和pandas处理csv文件中的时间方法
- 用python的pandas库对CSV格式的文件进行简单的统计分析操作
- PHP按行读取、处理较大CSV文件的代码实例
- Python使用pandas处理CSV文件的实例讲解
- 利用Pandas库读取CSV文件
- Python pandas读取csv文件直接转化成二维列表(Dataframe转化为list)
