聚类分析(K-means 层次聚类和基于密度DBSCAN算法三种实现方式)
2018-08-09 13:53
603 查看
之前也做过聚类,只不过是用经典数据集,这次是拿的实际数据跑的结果,效果还可以,记录一下实验过程。
首先:
确保自己数据集是否都完整,不能有空值,最好也不要出现为0的值,会影响聚类的效果。
其次:
想好要用什么算法去做,K-means,层次聚类还是基于密度算法,如果对这些都不算特别深入了解,那就都尝试一下吧,我就是这样做的。
好了,简单开始讲解实验的过程吧。
第一步
环境:
Windows10 + Anaconda3 + Pycharm
为了避免中文编码错误,在python文件上加一句中文编码设置代码:
#-*- coding:utf-8 -*-
并且在Pycharm下修改编码设置,安心。方法如下:
在Pycharm下打开你的项目,左上角File–>Settings–>Editor–>File Encodings
点击那个+号先把你的项目添加进来,然后按照上面截图将编码格式改成UTF-8,点击Apply,再点OK。这样编码错误就不会出现了,如果读入的文件含有中文还是出现编码错误,那就是你保存的文件编码格式不对,去修改文件保存的编码格式,自行百度。
一些库的准备:
import pandas as pd from sklearn.cluster import KMeans #导入K均值聚类算法 from sklearn.cluster import AgglomerativeClustering #层次聚类 from sklearn.cluster import DBSCAN #具有噪声的基于密度聚类方法 import matplotlib.pyplot as plt #matplotlib画图 from sklearn.decomposition import PCA #主成分分析 from mpl_toolkits.mplot3d import Axes3D #画三维图
这些库都有注释,想了解详细自己去看官方文档。
第二步
贴上了完整的代码,只需要改文件路径就可以了。
#-*- coding:utf-8 -*-
import pandas as pd
from sklearn.cluster import KMeans #导入K均值聚类算法
from sklearn.cluster import AgglomerativeClustering #层次聚类
from sklearn.cluster import DBSCAN #具有噪声的基于密度聚类方法
import matplotlib.pyplot as plt #matplotlib画图
from sklearn.decomposition import PCA #主成分分析
from mpl_toolkits.mplot3d import Axes3D #画三维图
pltcolor=['b<', 'g>', 'r1', 'c2', 'm3', 'y4', 'ks', 'wp'] #颜色
inputfile = 'E:/Work/python/data.xlsx' #待聚类的数据文件,需要进行标准化的数据文件;
zscoredfile = 'E:/Work/python/zdata.xlsx' #标准差计算后的数据存储路径文件;
data = pd.read_excel(inputfile) #读取数据,因为我的文件时CSV格式的,直接read_excel就可以,不是的话自己写一个文件读取的方法读进来。
iteration = 5000 # 聚类最大循环数
d = []
mink = 4 #聚类的类别范围K值下界
maxk = mink + 1 #聚类的列别范围上界
#data = (data - data.mean(axis = 0))/(data.std(axis = 0)) #简洁的语句实现了标准化变换,类似地可以实现任何想要的变换。也可以直接在原始数据里面就先处理好,这句话就可以不用了。
#data.columns=['Z'+i for i in data.columns] #表头重命名。
#data.to_excel(zscoredfile, index = False) #数据写入,标准化之后数据重新写入,直接覆盖掉之前的文件里的内容
# svd_solver : string {'auto', 'full', 'arpack', 'randomized'} 这个参数可以在PCA算法里看到
pca = PCA(n_components=2, svd_solver='full') # 输出两维 PCA主成分分析抽象出维度
#下面注释的几行是其它几种PCA主成分分析方法,可以尝试使用。
# from sklearn.decomposition import KernelPCA
# kernel : "linear" | "poly" | "rbf" | "sigmoid" | "cosine" | "precomputed"
# pca = KernelPCA(n_components = 2, kernel = "poly")
# method : {'lars', 'cd'}
# from sklearn.decomposition import SparsePCA
# pca = SparsePCA(n_components = 2, method = 'cd')
# from sklearn.decomposition import TruncatedSVD
# algorithm : string, default = "randomized" "arpack"
# pca = TruncatedSVD(algorithm = "arpack")
newData = pca.fit_transform(data) # 载入N维
if __name__ == '__main__' :
print("Kmeans")
for k in range(mink, maxk): # k取值在mink-maxk值之间,做kmeans聚类,看不同k值对应的簇内误差平方和
outputfile = 'E:/Work/python/fenlei%d.xlsx' % k
# 读取数据并进行聚类分析
# 调用k-means算法,进行聚类分析
kmodel = KMeans(n_clusters=k, init='k-means++', n_jobs=5, max_iter=iteration) # n_jobs是并行数,一般等于CPU数较好,max_iter是最大迭代次数,init='K-means++'参数的设置可以让初始化均值向量的时候让这几个簇中心尽量分开
kmodel.fit(data) # 训练模型
r1 = pd.Series(kmodel.labels_).value_counts() # 统计各个类别的数目
#r2 = pd.DataFrame(kmodel.cluster_centers_) # 找出聚类中心
print(r1)
kmlabels = kmodel.labels_ # 得到类别,label_ 是内部变量
#print(pd.Series(kmlabels, index=data.index)) #输出为序号和对应的类别,以序号为拼接列,把类别加到最后面一列
r = pd.concat([data, pd.Series(kmlabels, index=data.index)], axis=1) # 详细输出每个样本对应的类别,横向连接(0是纵向),得到聚类中心对应的类别下的数目
r.columns = list(data.columns) + [u'聚类类别'] # 重命名表头 加一列的表头
r.to_excel(outputfile) # 保存分类结果
d.append(kmodel.inertia_) # inertia簇内误差平方和
if mink >= maxk - 1:
x = []
y = []
for i in range(0, len(r1)):
x.append([])
y.append([])
for i in range(0, len(kmlabels)):
labelnum = kmlabels[i] #输出每条数据的类别标签
if labelnum >= 0:
x[labelnum].append(newData[i][0])
y[labelnum].append(newData[i][1])
# blue,green,red,cyan,magenta,yellow,black,white
for i in range(0, len(r1)):
plt.plot(x[i], y[i], pltcolor[i])
plt.show()
if mink < maxk - 1 :
plt.plot(range(mink, maxk), d, marker='o')
plt.xlabel('number of clusters')
plt.ylabel('distortions')
plt.show()
'''
k = 5 #需要进行的聚类类别数
outputfile = 'E:/Work/python/fenlei%d.xlsx' % k
iteration = 500 #聚类最大循环数
#读取数据并进行聚类分析
data = pd.read_excel(inputfile) #读取数据
#调用k-means算法,进行聚类分析
kmodel = KMeans(n_clusters = k, n_jobs = 3, max_iter=iteration) #n_jobs是并行数,一般等于CPU数较好
kmodel.fit(data) #训练模型
r1 = pd.Series(kmodel.labels_).value_counts() #统计各个类别的数目
r2 = pd.DataFrame(kmodel.cluster_centers_) #找出聚类中心
r = pd.concat([r2, r1], axis = 1) #横向连接(0是纵向),得到聚类中心对应的类别下的数目
r.columns = list(data.columns) + [u'类别数目'] #重命名表头
print (r)
r = pd.concat([data, pd.Series(kmodel.labels_, index = data.index)], axis = 1) #详细输出每个样本对应的类别
r.columns = list(data.columns) + [u'聚类类别'] #重命名表头
r.to_excel(outputfile) #保存分类结果
'''
if False:
def density_plot(data): #自定义作图函数
p = data.plot(kind='kde', linewidth = 2, subplots = True, sharex = False)
[p[i].set_ylabel('density') for i in range(k)]
plt.legend()
return plt
pic_output = 'E:/Work/python/' #概率密度图文件名前缀
for i in range(k):
print(u'%s%s.png' %(pic_output, i))
if i > 1:
density_plot(data[r[u'聚类类别']==i]).savefig(u'%s%s.png' %(pic_output, i))
if __name__ == "__main__2" :
print("AgglomerativeClustering")
for k in range(mink, maxk): # k取值1~11,做kmeans聚类,看不同k值对应的簇内误差平方和
outputfile = 'E:/Work/python/fenlei%d.xlsx' % k
# 读取数据并进行聚类分析
# 调用层次聚类算法,进行聚类分析
linkages = ['ward', 'average', 'complete']
kmodel = AgglomerativeClustering(linkage=linkages[2],n_clusters = k, affinity='manhattan')
kmodel.fit(data) # 训练模型
r1 = pd.Series(kmodel.labels_).value_counts() # 统计各个类别的数目
print(r1)
#r2 = pd.DataFrame(kmodel.cluster_centers_) # 找出聚类中心
kmlabels = kmodel.labels_
r = pd.concat([data, pd.Series(kmlabels, index=data.index)], axis=1) # 详细输出每个样本对应的类别,横向连接(0是纵向),得到聚类中心对应的类别下的数目
r.columns = list(data.columns) + [u'聚类类别'] # 重命名表头
#print(outputfile)
r.to_excel(outputfile) # 保存分类结果
if mink >= maxk - 1:
x = []
y = []
for i in range(0, len(r1)):
x.append([])
y.append([])
for i in range(0, len(kmlabels)):
labelnum = kmlabels[i]
if labelnum >= 0:
x[labelnum].append(newData[i][0])
y[labelnum].append(newData[i][1])
# blue,green,red,cyan,magenta,yellow,black,white
for i in range(0, len(r1)):
plt.plot(x[i], y[i], pltcolor[i])
plt.show()
if __name__ == "__main__3" :
print("DBSCAN")
outputfile = 'E:/Work/python/fenlei_DBSCAN.xlsx'
# 读取数据并进行聚类分析
# 调用DBSCAN算法,进行聚类分析
linkages = ['ward', 'average', 'complete']
#From scikit-learn: ['cityblock', 'cosine', 'euclidean', 'l1', 'l2', 'manhattan']. These metrics support sparse matrix inputs.
#From scipy.spatial.distance: ['braycurtis', 'canberra', 'chebyshev', 'correlation', 'dice', 'hamming', 'jaccard', 'kulsinski', 'mahalanobis', 'matching', 'minkowski', 'rogerstanimoto', 'russellrao', 'seuclidean', 'sokalmichener', 'sokalsneath', 'sqeuclidean', 'yule']
kmodel = DBSCAN(eps= 0.768, min_samples=160, n_jobs=7, metric='euclidean')
# kmodel = DBSCAN(eps=0.64, min_samples=100, n_jobs=7, metric='canberra')
kmodel.fit(data) # 训练模型
r1 = pd.Series(kmodel.labels_).value_counts() # 统计各个类别的数目
print(r1)
# r2 = pd.DataFrame(kmodel.cluster_centers_) # 找出聚类中心
kmlabels = kmodel.labels_
r = pd.concat([data, pd.Series(kmlabels, index=data.index)], axis=1) # 详细输出每个样本对应的类别, 横向连接(0是纵向),得到聚类中心对应的类别下的数目
r.columns = list(data.columns) + [u'聚类类别'] # 重命名表头
print(outputfile)
r.to_excel(outputfile) # 保存分类结果
rlen = len(r1)
if (rlen <= 6):
x = []
y = []
for i in range(0, len(r1) + 1):
x.append([])
y.append([])
for i in range(0, len(kmlabels)):
labelnum = kmlabels[i] + 1
if labelnum >= 0:
x[labelnum].append(newData[i][0])
y[labelnum].append(newData[i][1])
# blue,green,red,cyan,magenta,yellow,black,white
for i in range(0, len(r1) + 1):
plt.plot(x[i], y[i], pltcolor[i])
plt.show()
第三步
可以运行看一下效果,下图是使用K-means聚类出来的效果,K值设为4:
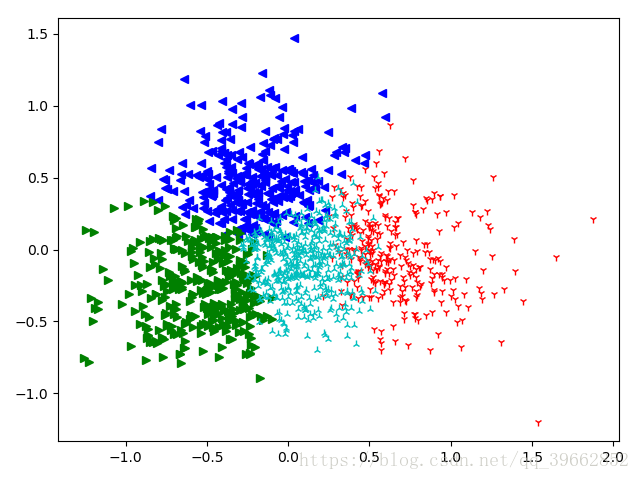
然后你可以去看输出文件分出的类别,可以尝试改变K值,直接改minK和maxK 的值就可以了。
相关文章推荐
- 机器学习知识点(十八)密度聚类DBSCAN算法Java实现
- Android HAL实现的三种方式(2) - 基于Service的HAL设计
- 基于密度的聚类算法(DBSCAN)的java实现
- 高效的六面体变换算法实现(二) —— 基于GPU的转换方式A
- 组合算法(三种实现方式)
- Android HAL实现的三种方式(2) - 基于Service的HAL设计
- Android HAL实现的三种方式(3) - 基于Manager的HAL设计
- 编写基于dbscan的GPS数据热点区域分析(二)算法的实现
- 数据挖掘-基于Kmeans算法、MBSAS算法及DBSCAN算法的newsgroup18828文本聚类器的JAVA实现(下)
- SCAN:基于密度的社团发现算法, SparklingGraph-实现PSCAN介绍
- 基于递归全排算法的最笨方式实现24点游戏
- 用户地理位置的聚类算法实现—基于DBSCAN和Kmeans的混合算法
- Android HAL实现的三种方式(1) - 基于JNI的简单HAL设计
- Android HAL实现的三种方式(1) - 基于JNI的简单HAL设计
- MySQL(一):分别基于mysqldump、lvm2、xtrabackup三种方式实现备份恢复
- 转载.Android HAL实现的三种方式(1) - 基于JNI的简单HAL设计
- Android HAL实现的三种方式(2) - 基于Service的HAL设计
- Android HAL实现的三种方式(2) - 基于Service的HAL设计
- 聚类 - 4 - 层次聚类、密度聚类(DBSCAN算法、密度最大值聚类)
- Android HAL实现的三种方式(3) - 基于Manager的HAL设计
