EM算法及OpenCV源码分析
2016-08-09 13:58
190 查看
关于EM原理,见:
http://blog.csdn.net/app_12062011/article/details/50350428
如果指定CvEM::START_E_STEP或CvEM::START_M_STEP参数,则不会出现同样的输入数据,得到不同结果的现象。如果指定CvEM::START_M_STEP参数,则以M步开始,
M步固定
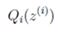
优化
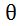
,必须给出概率
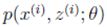
如果指定CvEM::START_E_STEP,则以E步开始,CvEMParams::means必须给出,CvEMParams::weights和CvEMParams::covs参数可给出可不给出,weights代表初始的各个成分的概率。
算法执行的终止条件。EM算法是迭代算法,自然终止条件可以是迭代次数达到了,或者两次迭代之间的差异小于epsilon就结束。
关于参数的解析,请参照机器学习中文参考手册。
emObj = EM //建立一个EM对象。
根据_params.start_step值执行不同过程,train,trainE和trainM。
这三个train过程都会返回logLikelihoods(Mat结构),_labels,probs(给定样本x属于各个类别的后验概率)。在训练之前,train函数里面会调用setTrainData准备训练数据,再调用do_train正式训练。setTrainData会做参数安全检查,如果是START_AUTO_STEP,则会打开K-means并把数据都转换成CV_32FC1。训练数据都保存到类成员trainSamples里。
执行do_train过程
1.clusterTrainSamples里调用kmeans方法聚类训练集成nclusters个类,并得到各个样本的类别。Kmeans执行前要保证数据是CV_32FC1类型,执行完后要转换成CV_64FC1类型。
2.根据labels将所有数据放到nclusters个矩阵中,分别计算每个矩阵的协方差矩阵和每个类别权值(该类样本数除以样本总数)。对每个协方差矩阵做奇异值分解(SVD),得到最大的奇异值的倒数。
3.反复循环执行E步,M步,直到满足如下条件:
trainLogLikelihoodDelta 表示两次相邻迭代过程中对数似然概率的增量。
E-step 源码:
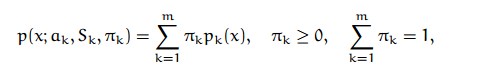
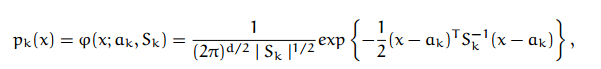
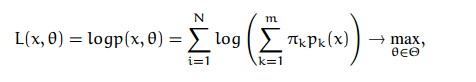
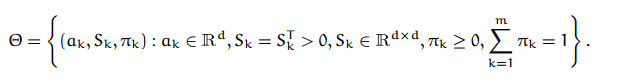
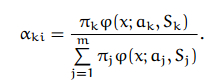
注意上面的公式,转换到log后,相乘除的变为相加减
M-step:

http://blog.csdn.net/app_12062011/article/details/50350428
EM算法的启动和终止
算法执行的开始步骤有三种指定方式。如果使用了CvEM::START_AUTO_STEP,则会调用k-means算法估计最初的参数,K-means会随机地初始化类中心,KMEANS_PP_CENTERS,这会导致EM算法得到不同的结果,如果数据量越大,则这种差异性会变小。如果指定CvEM::START_E_STEP或CvEM::START_M_STEP参数,则不会出现同样的输入数据,得到不同结果的现象。如果指定CvEM::START_M_STEP参数,则以M步开始,
M步固定
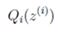
优化
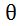
,必须给出概率
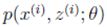
如果指定CvEM::START_E_STEP,则以E步开始,CvEMParams::means必须给出,CvEMParams::weights和CvEMParams::covs参数可给出可不给出,weights代表初始的各个成分的概率。
算法执行的终止条件。EM算法是迭代算法,自然终止条件可以是迭代次数达到了,或者两次迭代之间的差异小于epsilon就结束。
关于参数的解析,请参照机器学习中文参考手册。
CvEM::train函数中执行以下过程:
init_params。emObj = EM //建立一个EM对象。
根据_params.start_step值执行不同过程,train,trainE和trainM。
这三个train过程都会返回logLikelihoods(Mat结构),_labels,probs(给定样本x属于各个类别的后验概率)。在训练之前,train函数里面会调用setTrainData准备训练数据,再调用do_train正式训练。setTrainData会做参数安全检查,如果是START_AUTO_STEP,则会打开K-means并把数据都转换成CV_32FC1。训练数据都保存到类成员trainSamples里。
执行do_train过程
1.clusterTrainSamples里调用kmeans方法聚类训练集成nclusters个类,并得到各个样本的类别。Kmeans执行前要保证数据是CV_32FC1类型,执行完后要转换成CV_64FC1类型。
2.根据labels将所有数据放到nclusters个矩阵中,分别计算每个矩阵的协方差矩阵和每个类别权值(该类样本数除以样本总数)。对每个协方差矩阵做奇异值分解(SVD),得到最大的奇异值的倒数。
3.反复循环执行E步,M步,直到满足如下条件:
trainLogLikelihoodDelta 表示两次相邻迭代过程中对数似然概率的增量。
E-step 源码:
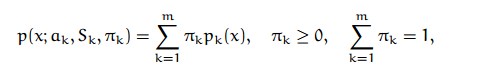
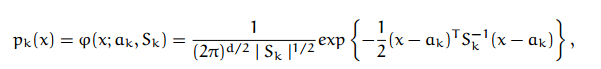
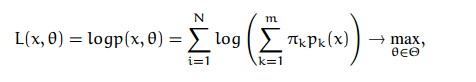
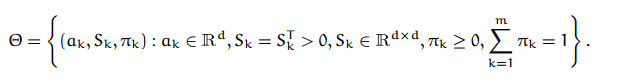
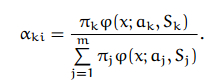
注意上面的公式,转换到log后,相乘除的变为相加减
Vec2d EM::computeProbabilities(const Mat& sample, Mat* probs) const
{
// L_ik = log(weight_k) - 0.5 * log(|det(cov_k)|) - 0.5 *(x_i - mean_k)' cov_k^(-1) (x_i - mean_k)]
// q = arg(max_k(L_ik))
// probs_ik = exp(L_ik - L_iq) / (1 + sum_j!=q (exp(L_ij - L_iq))
// see Alex Smola's blog http://blog.smola.org/page/2 for
// details on the log-sum-exp trick
CV_Assert(!means.empty());
CV_Assert(sample.type() == CV_64FC1);
CV_Assert(sample.rows == 1);
CV_Assert(sample.cols == means.cols);
int dim = sample.cols;
Mat L(1, nclusters, CV_64FC1); //L 1*nclusters
int label = 0;
for(int clusterIndex = 0; clusterIndex < nclusters; clusterIndex++)
{
const Mat centeredSample = sample - means.row(clusterIndex); //减去均值
Mat rotatedCenteredSample = covMatType != EM::COV_MAT_GENERIC ?
centeredSample : centeredSample * covsRotateMats[clusterIndex];
double Lval = 0;
for(int di = 0; di < dim; di++)
{
double w = invCovsEigenValues[clusterIndex].at<double>(covMatType != EM::COV_MAT_SPHERICAL ? di : 0);
//对角线上的值或者每行第一个值
double val = rotatedCenteredSample.at<double>(di);
Lval += w * val * val;//方差乘以权值 协方差矩阵的倒数的平方
}
CV_DbgAssert(!logWeightDivDet.empty());
L.at<double>(clusterIndex) = logWeightDivDet.at<double>(clusterIndex) - 0.5 * Lval;
// note: logWeightDivDet = log(weight_k) - 0.5 * log(|det(cov_k)|)
// note: L.at<double>(clusterIndex) = log(weight_k) - 0.5 * log(|det(cov_k)|)-0.5 * Lval
if(L.at<double>(clusterIndex) > L.at<double>(label))
label = clusterIndex; //求似然最大的label值
}
double maxLVal = L.at<double>(label); //
Mat expL_Lmax = L; // exp(L_ij - L_iq) //L 1*nclusters
for(int i = 0; i < L.cols; i++)
expL_Lmax.at<double>(i) = std::exp(L.at<double>(i) - maxLVal);
double expDiffSum = sum(expL_Lmax)[0]; // sum_j(exp(L_ij - L_iq))
if(probs) //probs
{
probs->create(1, nclusters, CV_64FC1);
double factor = 1./expDiffSum;
expL_Lmax *= factor;
expL_Lmax.copyTo(*probs);
}
Vec2d res;
res[0] = std::log(expDiffSum) + maxLVal - 0.5 * dim * CV_LOG2PI; //dim样本维数 CV_LOG2PI (1.8378770664093454835606594728112)
//
res[1] = label;
return res;
}M-step:

相关文章推荐
- 【OpenCV】SIFT原理与源码分析:DoG尺度空间构造
- 【OpenCV】SIFT原理与源码分析:关键点搜索与定位
- SIFT算法原理与OpenCV源码分析6:OpenCV实现SIFT算法,特征检测器FeatureDetector
- opencv源码解析之(6):hog源码分析
- opencv源码解析之(6):hog源码分析 -http://www.cnblogs.com/tornadomeet
- OpenCV深入学习(8)--calcHist源码分析
- 【OpenCV】SIFT原理与源码分析
- 【OpenCV】SIFT原理与源码分析
- SIFT算法原理与OpenCV源码分析4:方向赋值
- opencv源码解析之:hog源码分析
- 【OpenCV】SIFT原理与源码分析:方向赋值
- OpenCV学习笔记(29)KAZE 算法原理与源码分析(三)特征检测与描述
- 【OpenCV】SIFT原理与源码分析:DoG尺度空间构造
- 【OpenCV】SIFT原理与源码分析:DoG尺度空间构造
- SIFT算法原理与OpenCV源码分析5:关键点描述
- 【OpenCV】SIFT原理与源码分析:DoG尺度空间构造
- 【OpenCV】SIFT原理与源码分析
- OpenCV学习笔记(27)KAZE 算法原理与源码分析(一)非线性扩散滤波
- 【OpenCV】SIFT原理与源码分析:DoG尺度空间构造
- opencv 仿射变换 计算旋转矩阵源码分析
