fastqc, Per Base Sequence Content
2015-11-09 15:23
651 查看
Per Base Sequence Content
对所有reads的每一个位置,统计ATCG四种碱基(正常情况)的分布:
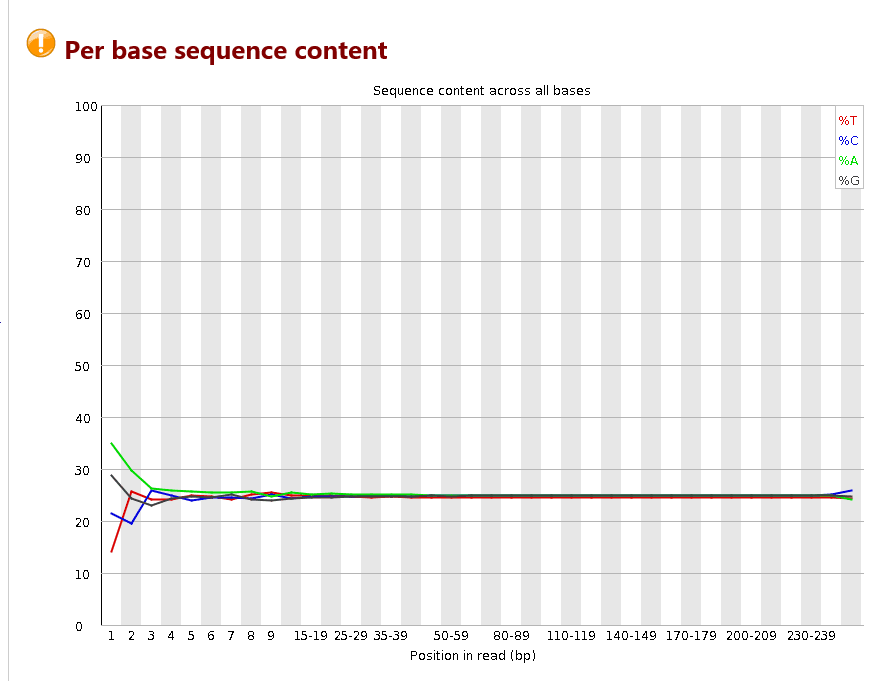
横轴为位置,纵轴为百分比。 正常情况下四种碱基的出现频率应该是接近的,而且没有位置差异。因此好的样本中四条线应该平行且接近。当部分位置碱基的比例出现bias时,即四条线在某些位置纷乱交织,往往提示我们有overrepresented sequence的污染。当所有位置的碱基比例一致的表现出bias时,即四条线平行但分开,往往代表文库有bias (建库过程或本身特点),或者是测序中的系统误差。
当任一位置的A/T比例与G/C比例相差超过10%,报"WARN";当任一位置的A/T比例与G/C比例相差超过20%,报"FAIL"。
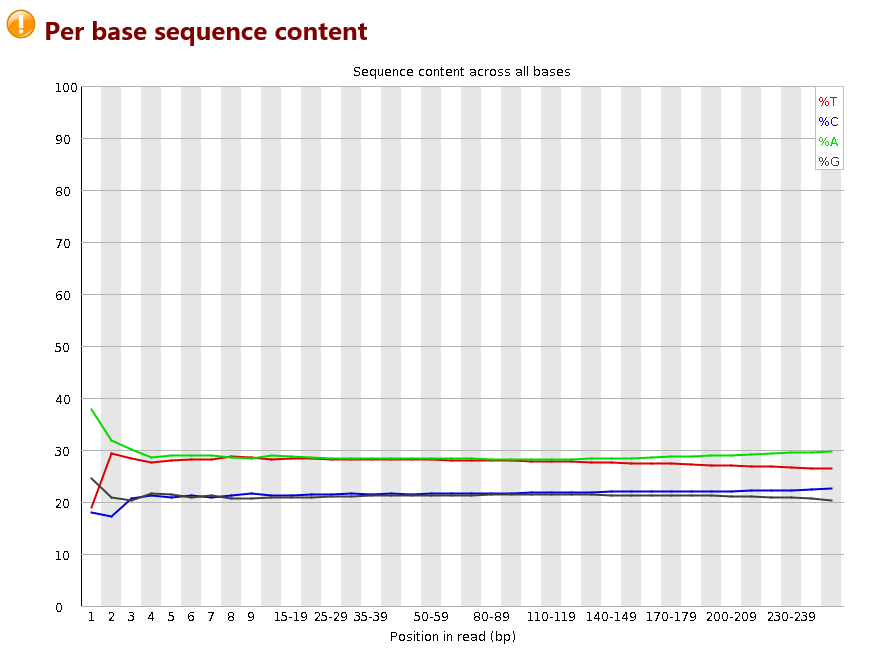
第一个图其实是不正常的,第二个图才是正常的,因为植物基因组,本来AT就比GC要高,第一个图之所以都一样,是因为有细菌或者叶绿体污染,导致
四种碱基含量差不多了。。。
中心测序,每次都是前面会有波动,可能是由于刚开始要测接头,可能要切完以后,信号不稳定等因素~~
freemao
FAFU
对所有reads的每一个位置,统计ATCG四种碱基(正常情况)的分布:

横轴为位置,纵轴为百分比。 正常情况下四种碱基的出现频率应该是接近的,而且没有位置差异。因此好的样本中四条线应该平行且接近。当部分位置碱基的比例出现bias时,即四条线在某些位置纷乱交织,往往提示我们有overrepresented sequence的污染。当所有位置的碱基比例一致的表现出bias时,即四条线平行但分开,往往代表文库有bias (建库过程或本身特点),或者是测序中的系统误差。
当任一位置的A/T比例与G/C比例相差超过10%,报"WARN";当任一位置的A/T比例与G/C比例相差超过20%,报"FAIL"。

第一个图其实是不正常的,第二个图才是正常的,因为植物基因组,本来AT就比GC要高,第一个图之所以都一样,是因为有细菌或者叶绿体污染,导致
四种碱基含量差不多了。。。
中心测序,每次都是前面会有波动,可能是由于刚开始要测接头,可能要切完以后,信号不稳定等因素~~
freemao
FAFU
相关文章推荐
- UITableView左滑删除
- Android的线程使用来更新UI----Thread、Handler、Looper、TimerTask等
- uiautomator中文输入解决方案
- UITableView左滑出现多个按钮
- UITableView 类似通讯录的索引
- 禁止uiscrollview垂直方向滚动,只允许水平方向滚动;或只允许垂直方向滚动
- UIWindow的简单实用(二)
- 复古篇:重现iOS6UITableView的Group风格
- iOS-UIViewController创建的几种方法和UIWindow的介绍
- iOS-UIViewController创建的几种方法和UIWindow的介绍
- UIView的简单实用
- Android开发之利用Fragment建立动态UI
- UISegmentedControl
- 为什么在Pool中传递Queue对象无法运行
- UIMenuController的使用,对UILabel拷贝以及定制菜单 .posted on 2013-03-18 16:20
- UITableView代理方法不执行的原因
- iOS开发UI篇—CAlayer(自定义layer)
- UITableView删除多行
- MIUI 6 沉浸式状态栏调用方法
- UIlabel 属性text
