机器学习中随机森林、神经网络和xgboost的分类问题和判断分布的方法
一、
最近在做机器学习分类问题的评价,写了一些代码和评价方法
总的来说,用随机森林和其他分类器做好分类后对混淆矩阵进行处理可以得到rr和kappa系数,此外对于二分类变量,还可以计算出roc曲线和auc面积,在对随机森林的计算中,我得到了以下的代码:
library(randomForest)
library(moments)
library(car)
library("soiltexture")
library(caret)
setwd("d:/Z/test")
A<-matrix(0,30,1)
ALOW<-matrix(0,30,1)
AUP<-matrix(0,30,1)
KAPPA<-matrix(0,30,1)
trainall=read.csv("classT.csv")
trainall$veget <-as.factor(trainall$veget)
trainall$soilt <-as.factor(trainall$soilt)
trainall$lc <- as.factor(trainall$lc)
trainall$lcc<- as.factor(trainall$lcc)
trainall$geo <- as.factor(trainall$geo)
for (ttt in 1:30)
{
while(TRUE)
{
while(TRUE)
{
index<- sample(1:nrow(trainall), 449)
train<- trainall[index, ]
testdata<- trainall[-index, ]
train$class<- factor(train$class)
testdata$class<- factor(testdata$class)
if(length(levels(train$class))==10&&length(levels(testdata$class))==10)
break
}
TF=randomForest(class~.,data=train,mtry=5,ntree=1000,importance=T)
pre<-predict(TF,testdata,tpye=prob)
if(length(levels(pre))==10)
break
}
cc<-confusionMatrix(pre,testdata$class)
##cc$overall
a<-matrix(cc$overall)
A[ttt]=a[1,1]
KAPPA[ttt]=a[2,1]
ALOW[ttt]=a[3,1]
AUP[ttt]=a[4,1]
}
a1<-0
a2<-0
a3<-0
a4<-0
for(eee in 1:30)
{
a1<-a1+A[eee]
a2<-a2+KAPPA[eee]
a3<-a3+ALOW[eee]
a4<-a4+AUP[eee]
}
RESULT<-matrix(0,4,2)
RESULT[1,1]="Accuracy "
RESULT[2,1]="Kappa"
RESULT[3,1]="AccuracyLower"
RESULT[4,1]="AccuracyUpper"
RESULT[1,2]=a1/30
RESULT[2,2]=a2/30
RESULT[3,2]=a3/30
RESULT[4,2]=a4/30
write.table(RESULT, file ="d:/Z/test/RESULT1.txt", row.names = F, quote = F)
可以做30次交叉验证得出rr和kappa系数,但是最后结果得到的是直接的因子型变量,并不是我们要计算roc曲线而需要的prob,所以这种情况下不能得到roc曲线的计算,只用正确分类精度和混淆矩阵的kappa系数来评价分类器的好坏即可,在一些二分类变量中,我们计算出一些概率分布,我们可以通过r中以下的代码完成对roc和auc的计算:
library(pROC)
modelroc <- roc(newdata$y,pre)
plot(modelroc, print.auc=TRUE,auc.polygon=TRUE, grid=c(0.1, 0.2),
grid.col=c("green", "red"), max.auc.polygon=TRUE,
auc.polygon.col="skyblue", print.thres=TRUE)
######################
library(ROCR)
pred <- prediction(pre,newdata$y)
performance(pred,'auc')@y.values #AUC值
perf <- performance(pred,'tpr','fpr')
plot(perf)
###########
roc(myData$label, myData$score, plot=TRUE,print.thres=TRUE, print.auc=TRUE)
###############
roc1 <- roc(myData$label, myData$score)
roc2 <- roc(myData2$label,myData2$score)
plot(roc1, col="blue")
plot.roc(roc2, add=TRUE,col="red")
具体方法参考cran pROC
二、在对xgboost的分类问题中遇到了一些问题,
library(xgboost)
library(moments)
library(car)
library(sp)
library("soiltexture")
setwd("d:/Z/test")
trainall=read.csv("classTF.csv")
trainall=read.csv("classT.csv")
index<- sample(1:nrow(trainall), 449)
train<- trainall[index, ]
testdata<- trainall[-index, ]
##############data.matrix(X_test[,-1]))
ttt<-as.matrix(train)
tt<-as.matrix(testdata)
TF=xgboost(data=ttt[,-19],label=train$sand,max.depth= 2, eta = 1, nthread = 2, nround = 2, objective = "binary:logistic")
TF=xgboost(data=ttt[,-19],label=train$sand,max.depth= 10, eta = 1, nthread = 2, nround = 10)
TF=xgboost(data=ttt[,-19],label=train$sand,max.depth= 10, subsample=1,colsample_bytree=0.8,,min_child_weight=0.8,eta = 1, nthread =2, nround = 10)
#######TF=xgboost(data=ttt[,-19],label=train$sand,max.depth= 10, subsample=1,min_child_weight=0.8,colsample_bytree=0.8,eta = 1, nthread =2, nround = 10)
TF=xgboost(data=ttt,label=train$sand,max.depth= 2, eta = 1, nthread = 2, nround = 2)
importance_matrix <- xgb.importance(TF)
TF=xgboost(data=ttt[,-19],label=train$sand,max.depth= 5, eta = 1, nthread = 2, nround = 2)
pre<-predict(TF,tt[,-19])
apre<-as.matrix(pre)
att<-as.matrix(testdata$sand)
c<-cbind(apre,att)
mse<-0
for(i in 1:193)
{
mse<-mse+(att[i]-apre[i])*(att[i]-apre[i])
}
sqrt(mse/193)
rr1<-0
rr2<-0
rr3<-0
for(i in 1:193)
{
rr1<-rr1+att[i]
}
rr1<-rr1/193
for(j in 1:193)
{
rr2<-rr2+(apre[j]-att[j])*(apre[j]-att[j])
}
for(k in 1:193)
{
rr3<-rr3+(att[k]-rr1)*(att[k]-rr1)
}
RR<-(1-rr2/rr3)*100
经过此代码的结果并没有提高的很多,反而会有一些下降,主题是参数的调整问题,还有就是数据在这个算法的放置问题,不知道是不是与随机森林的不同(rf),迭代的次数没有设置好,问题与神经网络类似,得到的预测数值有很多重复值。这是一个不好的预测,表明了环境变量没有很好的应用在这个模型上。对于出图的程序也存在这个问题。
接下来将对xgboost的参数和变量的加入进行一些变化的调整,与rf包和ranger包进行对比。
三、
对于glm的回归预测模型,效果不好的原因(40%rr,16rmse)
library(xgboost)
library(moments)
library(car)library(sp)
library("soiltexture")
library(stats)
setwd("d:/Z/test")
RMSE<-matrix(0,30,1)
RR<-matrix(0,30,1)
for(ii in 1:30)
{
trainall=read.csv("train642.csv")
index<- sample(1:nrow(trainall), 449)
train<- trainall[index, ]
t2<- trainall[-index, ]
TF1<-glm(ILR_1~veget+geo+soilt+lc+aspect+slope+curvature+rain+tem+dem+soc+ndvi+conindex+lon+lat+plan+profile+vellyd+bi+savi+rain1,data=train,family=gaussian())
TF2<-glm(ILR_2~veget+geo+soilt+lc+aspect+slope+curvature+rain+tem+dem+soc+ndvi+conindex+lon+lat+plan+profile+vellyd+bi+savi+rain1,data=train,family=gaussian())
ILR_1.pre<-predict(TF1,t2)
ILR_2.pre<-predict(TF2,t2)
RF.prediction<-matrix(0,193,2)
RF.prediction[,1]<-ILR_1.pre
RF.prediction[,2]<-ILR_2.pre
pred.Sand =100/(1+exp(sqrt(2)*RF.prediction[,1])+sqrt(exp(sqrt(2)*RF.prediction[,1]))*exp(sqrt(3/2)*RF.prediction[,2]))
apre<-as.matrix(pred.Sand)
att<-as.matrix(t2$sand)
c<-cbind(apre,att)
mse<-0
for(i in 1:193)
{
mse<-mse+(att[i]-apre[i])*(att[i]-apre[i])
}
RMSE[ii]<-sqrt(mse/193)
rr1<-0
rr2<-0
rr3<-0
for(i in 1:193)
{
rr1<-rr1+att[i]
}
rr1<-rr1/193
for(j in 1:193)
{
rr2<-rr2+(apre[j]-att[j])*(apre[j]-att[j])
}
for(k in 1:193)
{
rr3<-rr3+(att[k]-rr1)*(att[k]-rr1)
}
RR[ii]<-(1-rr2/rr3)*100
ii<ii+1
}
RR
RMSE
rrr<-0
rmsee<-0
for(i in 1:30)
{
rrr<-rrr+RR[i]
rmsee<-rmsee+RMSE[i]
}
rr<-rrr/30
rmse<-rmsee/30
对于以上的程序中,我得到了rr和rmse的值,分别为40%和16
与回归的rf相比,这是不好的(50%,14),我们注意到在公式中的glm选择的方法为高斯,即需要样本符合正态分布才可以,但是我们通过这样的一个r语言包:
library(fitdistrplus)
可以得到一个未知分布的分布情况,并根据最小的误差选择出最符合这个数据的分布。
x <- rnorm(642)
这是一个正态分布的数据,我们可以容易的看出这个程序给出的最接近分布。
但是我们的数据在整个流域中并不是一个正态分布
此外的两种检验方法:
1.
sand<-train$sand
ilr<-train$ILR_1
x <- rnorm(642)
x2 <- rnorm(642)
ks.test(x, x2)
ks.test(x, 'pnorm')
ks.test(ilr, 'pnorm')
ks.test(sand, 'pnorm')
ks.test(x, 'pexp')
2.
x <- rnorm(642)
x2 <- rnorm(642)
shapiro.test(x2)
chisq.test(sand)
回到我们的数据问题,我们又原始数据sand’和对数转化数据两种,我们分别对他们的数据分布类型做图:
Sand:
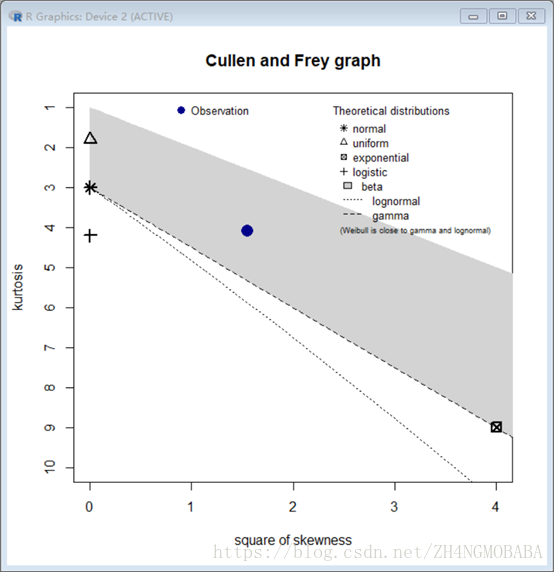
统计为:
min: 0.98 max: 99.66
median: 25.06398
mean: 30.57073
estimated sd: 21.96976
estimated skewness: 1.246292
estimated kurtosis: 4.079616
Ilr为:
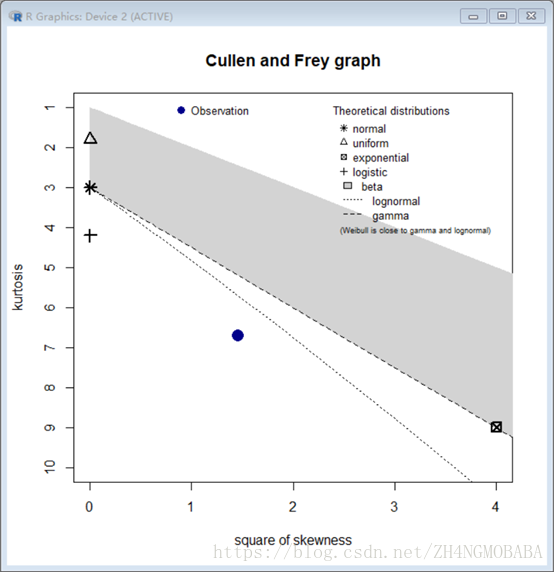
统计为;
min: -4.506902 max: 2.968629
median: 0.6145865
mean: 0.5308409
estimated sd: 0.924625
estimated skewness: -1.206198
estimated kurtosis: 6.71577
我们可以看到我们的数据sand应该是gamma或者beta分布的
Ilr应该是对数正态分布,而不是正态分布
阅读更多- 【机器学习】神经网络(一)——多类分类问题
- 机器学习:神经网络、正则化、多分类问题与Python代码实现
- 随机森林分类和adaboost分类方法的异同之处
- XGBoost:多分类问题
- XGBoost解决多分类问题
- 【HowTo ML】分类问题->神经网络入门
- 机器学习—第四周—作业2—用深度神经网络分类图像
- Spark2.0机器学习系列之7:多类分类问题(方法归总和分类结果评估)
- 【机器学习】随机初始化思想神经网络总结
- 数据挖掘学习------------------4-分类方法-4-神经网络(ANN)
- 机器学习-分类问题评估方法
- 【机器学习】动手写一个全连接神经网络(三):分类
- 利用随机森林,xgboost,logistic回归,预测泰坦尼克号上面的乘客的获救概率
- 以垃圾邮件判定方法探索机器学习中的二分类判定问题
- 机器学习之文本分类-从词频统计到神经网络(二)
- TensorFlow训练神经网络解决二分类问题
- Spark2.0机器学习系列之8:多类分类问题(方法归总和分类结果评估)
- 机器学习实验报告:利用3层神经网络对CIFAR-10图像数据库进行分类
- 【机器学习】【神经网络与深度学习】不均匀正负样本分布下的机器学习 《讨论集》
- XGBoost:二分类问题
