朴素贝叶斯分类器简介及C++实现(性别分类)
2017-09-22 16:25
281 查看
贝叶斯分类器是一种基于贝叶斯定理的简单概率分类器。
在机器学习中,朴素贝叶斯分类器是一系列以假设特征之间强(朴素)独立下运用贝叶斯定理为基础的简单概率分类器。朴素贝叶斯是文本分类的一种热门(基准)方法,文本分类是以词频为特征判断文件所属类别或其它(如垃圾邮件、合法性、体育或政治等等)的问题。通过适当的预处理,它可以与这个领域更先进的方法(包括支持向量机)相竞争。
朴素贝叶斯分类器是高度可扩展的,因此需要数量与学习问题中的变量(特征/预测器)成线性关系的参数。
在统计学和计算机科学文献中,朴素贝叶斯模型有各种名称,包括简单贝叶斯和独立贝叶斯。所有这些名称都参考了贝叶斯定理在该分类器的决策规则中的使用,但朴素贝叶斯不一定用到贝叶斯方法。
朴素贝叶斯是一种构建分类器的简单方法。该分类器模型会给问题实例分配用特征值表示的类标签,类标签取自有限集合。它不是训练这种分类器的单一算法,而是一系列基于相同原理的算法:所有朴素贝叶斯分类器都假定样本每个特征与其它特征都不相关。举个例子,如果一种水果其具有红,圆,直径大概3英寸等特征,该水果可以被判定为是苹果。尽管这些特征相互依赖或者有些特征由其他特征决定,然而朴素贝叶斯分类器认为这些属性在判定该水果是否为苹果的概率分布上独立的。
对于某些类型的概率模型,在监督式学习的样本集中能获取得非常好的分类效果。在许多实际应用中,朴素贝叶斯模型参数估计使用最大似然估计方法;换而言之,在不用到贝叶斯概率或者任何贝叶斯模型的情况下,朴素贝叶斯模型也能奏效。
朴素贝叶斯分类器的一个优势在于只需要根据少量的训练数据估计出必要的参数(变量的均值和方差)。由于变量独立假设,只需要估计各个变量的方法,而不需要确定整个协方差矩阵。
朴素贝叶斯概率模型:理论上,概率模型分类器是一个条件概率模型:p(C|F1,…,Fn)。独立的类别变量C有若干类别,条件依赖于若干特征变量F1,F2,…,Fn。但问题在于如果特征数量n较大或者每个特征能取大量值时,基于概率模型列出概率表变得不现实。所以我们修改这个模型使之变得可行。贝叶斯定理有以下式子:
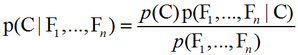
用朴素的语言可以表达为:
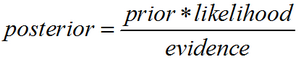
实际中,我们只关心分式中的分子部分,因为分母不依赖于C而且特征Fi的值是给定的,于是分母可以认为是一个常数。这样分子就等价于联合分布(在概率论中,对两个随机变量X和Y,其联合分布是同时对于X和Y的概率分布。对离散随机变量而言,联合分布概率质量函数为Pr(X=x& Y=y),即P(X=x and Y=y)=P(Y=y|X=x)P(X=x)=P(X=x|Y=y)P(Y=y))模型:p(C,F1,…,Fn)。重复使用链式法则(chain rule,是求复合函数导数的一个法则),可将该式写成条件概率(conditional probability,就是事件A在另外一个事件B已经发生条件下的发生概率,条件概率表示为P(A|B),读作”在B条件下A的概率”)的形式,如下所示:

其中Z(证据因子)是一个只依赖于F1,…,Fn等的缩放因子,当特征变量的值已知时是一个常数。由于分解成所谓的类先验概率p(C)和独立概率分布p(Fi|C),上述概率模型的可掌控性得到很大的提高。如果这是一个k分类问题,且每个p(Fi|C=c)可以表达为r个参数,于是相应的朴素贝叶斯模型有(k-1)+nrk个参数。实际应用中,通常取k=2(二分类问题),r=1(伯努利分布作为特征),因此模型的参数个数为2n+1,其中n是二值分类特征的个数。 从概率模型中构造分类器:以上导出了独立分布特征模型,也就是朴素贝叶斯概率模型。朴素贝叶斯分类器包括了这种模型和相应的决策规则。一个普通的规则就是选出最优可能的那个:这就是最大后验概率(MAP)决策准则。相应的分类器便是如下定义的classify公式:
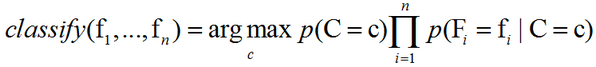
参数估计:所有的模型参数都可以通过训练集的相关频率来估计。常用方法是概率的最大似然估计。类的先验概率可以通过假设各类等概率来计算(先验概率 = 1 / (类的数量)),或者通过训练集的各类样本出现的次数来估计(A类先验概率=(A类样本的数量)/(样本总数))。为了估计特征的分布参数,我们要先假设训练集数据满足某种分布或者非参数模型。 高斯朴素贝叶斯:如果要处理的是连续数据,一种通常的假设是这些连续数值为高斯分布。例如,假设训练集中有一个连续属性x。我们首先对数据根据类别分类,然后计算每个类别中的x的均值和方差。令μc表示为x在c类上的均值,令σ2c为x类在c类上的方差。在给定类中某个值的概率P(x=v|c),可以通过将v表示为均值为μc,方差为σ2c正态分布计算出来,如下:
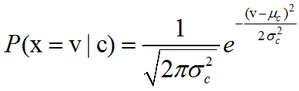
处理连续数值问题的另一种常用的技术是通过离散化连续数值的方法。通常,当训练样本数量较少或者是精确的分布已知时,通过概率分布的方法是一种更好的选择。在大量样本的情形下离散化的方法表现更优,因为大量的样本可以学习到数据的分布。由于朴素贝叶斯是一种典型的用到大量样本的方法(越大计算量的模型可以产生越高的分类精确度),所以朴素贝叶斯方法都用到离散化方法,而不是概率分布估计的方法。 样本修正:如果一个给定的类和特征值在训练集中没有一起出现过,那么基于频率的估计下该概率将为0。这将是一个问题。因为与其他概率相乘时将会把其他概率的信息统统去除。所以常常要求要对每个小类样本的概率估计进行修正,以保证不会出现有为0的概率出现。
尽管实际上独立假设常常是不准确的,但朴素贝叶斯分类器的若干特性让其在实践中能够取得令人惊奇的效果。特别地,各类条件特征之间的解耦意味着每个特征的分布都可以独立地被当做一维分布来估计。这样减轻了由于维数灾带来的阻碍,当样本的特征个数增加时就不需要使样本规模呈指数增长。然而朴素贝叶斯在大多数情况下不能对类概率做出非常准确的估计,但在许多应用中这一点并不要求。例如,朴素贝叶斯分类器中,依据最大后验概率决策规则只要正确类的后验概率比其他类要高就可以得到正确的分类。所以不管概率估计轻度的甚至是严重的不精确都不影响正确的分类结果。在这种方式下,分类器可以有足够的鲁棒性去忽略朴素贝叶斯概率模型上存在的缺陷。
以上内容主要摘自: 维基百科
以下code是根据维基百科中对性别分类的介绍实现的C++代码,最终结果与维基百科结果一致:
naive_bayes_classifier.hpp:
#ifndef FBC_NN_NAIVEBAYESCLASSIFIER_HPP_
#define FBC_NN_NAIVEBAYESCLASSIFIER_HPP_
#include <vector>
#include <tuple>
namespace ANN {
template<typename T>
struct sex_info { // height, weight, foot size, sex
T height;
T weight;
T foot_size;
int sex; // -1: unspecified, 0: female, 1: male
};
template<typename T>
struct MeanVariance { // height/weight/foot_size's mean and variance
T mean_height;
T mean_weight;
T mean_foot_size;
T variance_height;
T variance_weight;
T variance_foot_size;
};
// Gaussian naive Bayes
template<typename T>
class NaiveBayesClassifier {
public:
NaiveBayesClassifier() = default;
int init(const std::vector<sex_info<T>>& info);
int train(const std::string& model);
int predict(const sex_info<T>& info) const;
int load_model(const std::string& model) const;
private:
void calc_mean_variance(const std::vector<T>& data, std::tuple<T, T>& mean_variance) const;
T calc_attribute_probability(T value, T mean, T variance) const;
int store_model(const std::string& model) const;
MeanVariance<T> male_mv, female_mv;
std::vector<T> male_height, male_weight, male_foot_size;
std::vector<T> female_height, female_weight, female_foot_size;
T male_p = (T)0.5;
T female_p = (T)0.5;
int male_train_number = 0;
int female_train_number = 0;
};
} // namespace ANN
#endif // FBC_NN_NAIVEBAYESCLASSIFIER_HPP_ naive_bayes_classifier.cpp:#include "naive_bayes_classifier.hpp"
#include "common.hpp"
#include <math.h>
#include <iostream>
#include <algorithm>
#include <fstream>
namespace ANN {
template<typename T>
int NaiveBayesClassifier<T>::init(const std::vector<sex_info<T>>& info)
{
int length = info.size();
if (length < 2) {
fprintf(stderr, "train data length should be > 1: %d\n", length);
return -1;
}
male_train_number = 0;
female_train_number = 0;
for (int i = 0; i < length; ++i) {
if (info[i].sex == 0) {
++female_train_number;
female_height.push_back(info[i].height);
female_weight.push_back(info[i].weight);
female_foot_size.push_back(info[i].foot_size);
} else {
++male_train_number;
male_height.push_back(info[i].height);
male_weight.push_back(info[i].weight);
male_foot_size.push_back(info[i].foot_size);
}
}
male_p = (T)male_train_number / (male_train_number + female_train_number);
female_p = (T)female_train_number / (male_train_number + female_train_number);
return 0;
}
template<typename T>
int NaiveBayesClassifier<T>::train(const std::string& model)
{
std::tuple<T, T> mean_variance;
calc_mean_variance(male_height, mean_variance);
male_mv.mean_height = std::get<0>(mean_variance);
male_mv.variance_height = std::get<1>(mean_variance);
calc_mean_variance(male_weight, mean_variance);
male_mv.mean_weight = std::get<0>(mean_variance);
male_mv.variance_weight = std::get<1>(mean_variance);
calc_mean_variance(male_foot_size, mean_variance);
male_mv.mean_foot_size = std::get<0>(mean_variance);
male_mv.variance_foot_size = std::get<1>(mean_variance);
calc_mean_variance(female_height, mean_variance);
female_mv.mean_height = std::get<0>(mean_variance);
female_mv.variance_height = std::get<1>(mean_variance);
calc_mean_variance(female_weight, mean_variance);
female_mv.mean_weight = std::get<0>(mean_variance);
female_mv.variance_weight = std::get<1>(mean_variance);
calc_mean_variance(female_foot_size, mean_variance);
female_mv.mean_foot_size = std::get<0>(mean_variance);
female_mv.variance_foot_size = std::get<1>(mean_variance);
CHECK(store_model(model) == 0);
return 0;
}
template<typename T>
int NaiveBayesClassifier<T>::store_model(const std::string& model) const
{
std::ofstream file;
file.open(model.c_str(), std::ios::binary);
if (!file.is_open()) {
fprintf(stderr, "open file fail: %s\n", model.c_str());
return -1;
}
file.write((char*)&male_p, sizeof(male_p));
file.write((char*)&male_mv.mean_height, sizeof(male_mv.mean_height));
file.write((char*)&male_mv.mean_weight, sizeof(male_mv.mean_weight));
file.write((char*)&male_mv.mean_foot_size, sizeof(male_mv.mean_foot_size));
file.write((char*)&male_mv.variance_height, sizeof(male_mv.variance_height));
file.write((char*)&male_mv.variance_weight, sizeof(male_mv.variance_weight));
file.write((char*)&male_mv.variance_foot_size, sizeof(male_mv.variance_foot_size));
file.write((char*)&female_p, sizeof(female_p));
file.write((char*)&female_mv.mean_height, sizeof(female_mv.mean_height));
file.write((char*)&female_mv.mean_weight, sizeof(female_mv.mean_weight));
file.write((char*)&female_mv.mean_foot_size, sizeof(female_mv.mean_foot_size));
file.write((char*)&female_mv.variance_height, sizeof(female_mv.variance_height));
file.write((char*)&female_mv.variance_weight, sizeof(female_mv.variance_weight));
file.write((char*)&female_mv.variance_foot_size, sizeof(female_mv.variance_foot_size));
file.close();
return 0;
}
template<typename T>
int NaiveBayesClassifier<T>::predict(const sex_info<T>& info) const
{
T male_height_p = calc_attribute_probability(info.height, male_mv.mean_height, male_mv.variance_height);
T male_weight_p = calc_attribute_probability(info.weight, male_mv.mean_weight, male_mv.variance_weight);
T male_foot_size_p = calc_attribute_probability(info.foot_size, male_mv.mean_foot_size, male_mv.variance_foot_size);
T female_height_p = calc_attribute_probability(info.height, female_mv.mean_height, female_mv.variance_height);
T female_weight_p = calc_attribute_probability(info.weight, female_mv.mean_weight, female_mv.variance_weight);
T female_foot_size_p = calc_attribute_probability(info.foot_size, female_mv.mean_foot_size, female_mv.variance_foot_size);
T evidence = male_p * male_height_p * male_weight_p * male_foot_size_p +
female_p * female_height_p * female_weight_p * female_foot_size_p;
T male_posterior = male_p * male_height_p * male_weight_p * male_foot_size_p /*/ evidence*/;
T female_posterior = female_p * female_height_p * female_weight_p * female_foot_size_p /*/ evidence*/;
fprintf(stdout, "male posterior probability: %e, female posterior probability: %e\n",
male_posterior, female_posterior);
if (male_posterior > female_posterior) return 1;
else return 0;
}
template<typename T>
T NaiveBayesClassifier<T>::calc_attribute_probability(T value, T mean, T variance) const
{
return (T)1 / std::sqrt(2 * PI * variance) * std::exp(-std::pow(value - mean, 2) / (2 * variance));
}
template<typename T>
int NaiveBayesClassifier<T>::load_model(const std::string& model) const
{
std::ifstream file;
file.open(model.c_str(), std::ios::binary);
if (!file.is_open()) {
fprintf(stderr, "open file fail: %s\n", model.c_str());
return -1;
}
file.read((char*)&male_p, sizeof(male_p) * 1);
file.read((char*)&male_mv.mean_height, sizeof(male_mv.mean_height) * 1);
file.read((char*)&male_mv.mean_weight, sizeof(male_mv.mean_weight) * 1);
file.read((char*)&male_mv.mean_foot_size, sizeof(male_mv.mean_foot_size) * 1);
file.read((char*)&male_mv.variance_height, sizeof(male_mv.variance_height) * 1);
file.read((char*)&male_mv.variance_weight, sizeof(male_mv.variance_weight) * 1);
file.read((char*)&male_mv.variance_foot_size, sizeof(male_mv.variance_foot_size) * 1);
file.read((char*)&female_p, sizeof(female_p)* 1);
file.read((char*)&female_mv.mean_height, sizeof(female_mv.mean_height) * 1);
file.read((char*)&female_mv.mean_weight, sizeof(female_mv.mean_weight) * 1);
file.read((char*)&female_mv.mean_foot_size, sizeof(female_mv.mean_foot_size) * 1);
file.read((char*)&female_mv.variance_height, sizeof(female_mv.variance_height) * 1);
file.read((char*)&female_mv.variance_weight, sizeof(female_mv.variance_weight) * 1);
file.read((char*)&female_mv.variance_foot_size, sizeof(female_mv.variance_foot_size) * 1);
file.close();
return 0;
}
template<typename T>
void NaiveBayesClassifier<T>::calc_mean_variance(const std::vector<T>& data, std::tuple<T, T>& mean_variance) const
{
T sum{ 0 }, sqsum{ 0 };
for (int i = 0; i < data.size(); ++i) {
sum += data[i];
}
T mean = sum / data.size();
for (int i = 0; i < data.size(); ++i) {
sqsum += std::pow(data[i] - mean, 2);
}
// unbiased sample variances
T variance = sqsum / (data.size() - 1);
std::get<0>(mean_variance) = mean;
std::get<1>(mean_variance) = variance;
}
template class NaiveBayesClassifier<float>;
template class NaiveBayesClassifier<double>;
} // namespace ANN funset.cpp:#include "funset.hpp"
#include <iostream>
#include "perceptron.hpp"
#include "BP.hpp""
#include "CNN.hpp"
#include "linear_regression.hpp"
#include "naive_bayes_classifier.hpp"
#include "common.hpp"
#include <opencv2/opencv.hpp>
// ================================ naive bayes classifier =====================
int test_naive_bayes_classifier_train()
{
std::vector<ANN::sex_info<float>> info;
info.push_back({ 6.f, 180.f, 12.f, 1 });
info.push_back({5.92f, 190.f, 11.f, 1});
info.push_back({5.58f, 170.f, 12.f, 1});
info.push_back({5.92f, 165.f, 10.f, 1});
info.push_back({ 5.f, 100.f, 6.f, 0 });
info.push_back({5.5f, 150.f, 8.f, 0});
info.push_back({5.42f, 130.f, 7.f, 0});
info.push_back({5.75f, 150.f, 9.f, 0});
ANN::NaiveBayesClassifier<float> naive_bayes;
int ret = naive_bayes.init(info);
if (ret != 0) {
fprintf(stderr, "naive bayes classifier init fail: %d\n", ret);
return -1;
}
const std::string model{ "E:/GitCode/NN_Test/data/naive_bayes_classifier.model" };
ret = naive_bayes.train(model);
if (ret != 0) {
fprintf(stderr, "naive bayes classifier train fail: %d\n", ret);
return -1;
}
return 0;
}
int test_naive_bayes_classifier_predict()
{
ANN::sex_info<float> info = { 6.0f, 130.f, 8.f, -1 };
ANN::NaiveBayesClassifier<float> naive_bayes;
const std::string model{ "E:/GitCode/NN_Test/data/naive_bayes_classifier.model" };
int ret = naive_bayes.load_model(model);
if (ret != 0) {
fprintf(stderr, "load naive bayes classifier model fail: %d\n", ret);
return -1;
}
ret = naive_bayes.predict(info);
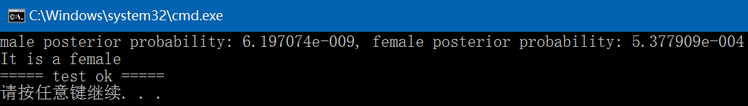
if (ret == 0) fprintf(stdout, "It is a female\n");
else fprintf(stdout, "It is a male\n");
return 0;
} 执行结果如下:
GitHub: https://github.com/fengbingchun/NN_Test
在机器学习中,朴素贝叶斯分类器是一系列以假设特征之间强(朴素)独立下运用贝叶斯定理为基础的简单概率分类器。朴素贝叶斯是文本分类的一种热门(基准)方法,文本分类是以词频为特征判断文件所属类别或其它(如垃圾邮件、合法性、体育或政治等等)的问题。通过适当的预处理,它可以与这个领域更先进的方法(包括支持向量机)相竞争。
朴素贝叶斯分类器是高度可扩展的,因此需要数量与学习问题中的变量(特征/预测器)成线性关系的参数。
在统计学和计算机科学文献中,朴素贝叶斯模型有各种名称,包括简单贝叶斯和独立贝叶斯。所有这些名称都参考了贝叶斯定理在该分类器的决策规则中的使用,但朴素贝叶斯不一定用到贝叶斯方法。
朴素贝叶斯是一种构建分类器的简单方法。该分类器模型会给问题实例分配用特征值表示的类标签,类标签取自有限集合。它不是训练这种分类器的单一算法,而是一系列基于相同原理的算法:所有朴素贝叶斯分类器都假定样本每个特征与其它特征都不相关。举个例子,如果一种水果其具有红,圆,直径大概3英寸等特征,该水果可以被判定为是苹果。尽管这些特征相互依赖或者有些特征由其他特征决定,然而朴素贝叶斯分类器认为这些属性在判定该水果是否为苹果的概率分布上独立的。
对于某些类型的概率模型,在监督式学习的样本集中能获取得非常好的分类效果。在许多实际应用中,朴素贝叶斯模型参数估计使用最大似然估计方法;换而言之,在不用到贝叶斯概率或者任何贝叶斯模型的情况下,朴素贝叶斯模型也能奏效。
朴素贝叶斯分类器的一个优势在于只需要根据少量的训练数据估计出必要的参数(变量的均值和方差)。由于变量独立假设,只需要估计各个变量的方法,而不需要确定整个协方差矩阵。
朴素贝叶斯概率模型:理论上,概率模型分类器是一个条件概率模型:p(C|F1,…,Fn)。独立的类别变量C有若干类别,条件依赖于若干特征变量F1,F2,…,Fn。但问题在于如果特征数量n较大或者每个特征能取大量值时,基于概率模型列出概率表变得不现实。所以我们修改这个模型使之变得可行。贝叶斯定理有以下式子:
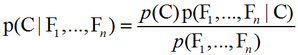
用朴素的语言可以表达为:
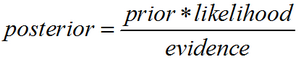
实际中,我们只关心分式中的分子部分,因为分母不依赖于C而且特征Fi的值是给定的,于是分母可以认为是一个常数。这样分子就等价于联合分布(在概率论中,对两个随机变量X和Y,其联合分布是同时对于X和Y的概率分布。对离散随机变量而言,联合分布概率质量函数为Pr(X=x& Y=y),即P(X=x and Y=y)=P(Y=y|X=x)P(X=x)=P(X=x|Y=y)P(Y=y))模型:p(C,F1,…,Fn)。重复使用链式法则(chain rule,是求复合函数导数的一个法则),可将该式写成条件概率(conditional probability,就是事件A在另外一个事件B已经发生条件下的发生概率,条件概率表示为P(A|B),读作”在B条件下A的概率”)的形式,如下所示:

其中Z(证据因子)是一个只依赖于F1,…,Fn等的缩放因子,当特征变量的值已知时是一个常数。由于分解成所谓的类先验概率p(C)和独立概率分布p(Fi|C),上述概率模型的可掌控性得到很大的提高。如果这是一个k分类问题,且每个p(Fi|C=c)可以表达为r个参数,于是相应的朴素贝叶斯模型有(k-1)+nrk个参数。实际应用中,通常取k=2(二分类问题),r=1(伯努利分布作为特征),因此模型的参数个数为2n+1,其中n是二值分类特征的个数。 从概率模型中构造分类器:以上导出了独立分布特征模型,也就是朴素贝叶斯概率模型。朴素贝叶斯分类器包括了这种模型和相应的决策规则。一个普通的规则就是选出最优可能的那个:这就是最大后验概率(MAP)决策准则。相应的分类器便是如下定义的classify公式:
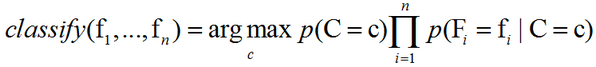
参数估计:所有的模型参数都可以通过训练集的相关频率来估计。常用方法是概率的最大似然估计。类的先验概率可以通过假设各类等概率来计算(先验概率 = 1 / (类的数量)),或者通过训练集的各类样本出现的次数来估计(A类先验概率=(A类样本的数量)/(样本总数))。为了估计特征的分布参数,我们要先假设训练集数据满足某种分布或者非参数模型。 高斯朴素贝叶斯:如果要处理的是连续数据,一种通常的假设是这些连续数值为高斯分布。例如,假设训练集中有一个连续属性x。我们首先对数据根据类别分类,然后计算每个类别中的x的均值和方差。令μc表示为x在c类上的均值,令σ2c为x类在c类上的方差。在给定类中某个值的概率P(x=v|c),可以通过将v表示为均值为μc,方差为σ2c正态分布计算出来,如下:
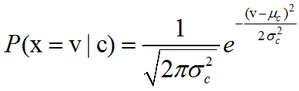
处理连续数值问题的另一种常用的技术是通过离散化连续数值的方法。通常,当训练样本数量较少或者是精确的分布已知时,通过概率分布的方法是一种更好的选择。在大量样本的情形下离散化的方法表现更优,因为大量的样本可以学习到数据的分布。由于朴素贝叶斯是一种典型的用到大量样本的方法(越大计算量的模型可以产生越高的分类精确度),所以朴素贝叶斯方法都用到离散化方法,而不是概率分布估计的方法。 样本修正:如果一个给定的类和特征值在训练集中没有一起出现过,那么基于频率的估计下该概率将为0。这将是一个问题。因为与其他概率相乘时将会把其他概率的信息统统去除。所以常常要求要对每个小类样本的概率估计进行修正,以保证不会出现有为0的概率出现。
尽管实际上独立假设常常是不准确的,但朴素贝叶斯分类器的若干特性让其在实践中能够取得令人惊奇的效果。特别地,各类条件特征之间的解耦意味着每个特征的分布都可以独立地被当做一维分布来估计。这样减轻了由于维数灾带来的阻碍,当样本的特征个数增加时就不需要使样本规模呈指数增长。然而朴素贝叶斯在大多数情况下不能对类概率做出非常准确的估计,但在许多应用中这一点并不要求。例如,朴素贝叶斯分类器中,依据最大后验概率决策规则只要正确类的后验概率比其他类要高就可以得到正确的分类。所以不管概率估计轻度的甚至是严重的不精确都不影响正确的分类结果。在这种方式下,分类器可以有足够的鲁棒性去忽略朴素贝叶斯概率模型上存在的缺陷。
以上内容主要摘自: 维基百科
以下code是根据维基百科中对性别分类的介绍实现的C++代码,最终结果与维基百科结果一致:
naive_bayes_classifier.hpp:
#ifndef FBC_NN_NAIVEBAYESCLASSIFIER_HPP_
#define FBC_NN_NAIVEBAYESCLASSIFIER_HPP_
#include <vector>
#include <tuple>
namespace ANN {
template<typename T>
struct sex_info { // height, weight, foot size, sex
T height;
T weight;
T foot_size;
int sex; // -1: unspecified, 0: female, 1: male
};
template<typename T>
struct MeanVariance { // height/weight/foot_size's mean and variance
T mean_height;
T mean_weight;
T mean_foot_size;
T variance_height;
T variance_weight;
T variance_foot_size;
};
// Gaussian naive Bayes
template<typename T>
class NaiveBayesClassifier {
public:
NaiveBayesClassifier() = default;
int init(const std::vector<sex_info<T>>& info);
int train(const std::string& model);
int predict(const sex_info<T>& info) const;
int load_model(const std::string& model) const;
private:
void calc_mean_variance(const std::vector<T>& data, std::tuple<T, T>& mean_variance) const;
T calc_attribute_probability(T value, T mean, T variance) const;
int store_model(const std::string& model) const;
MeanVariance<T> male_mv, female_mv;
std::vector<T> male_height, male_weight, male_foot_size;
std::vector<T> female_height, female_weight, female_foot_size;
T male_p = (T)0.5;
T female_p = (T)0.5;
int male_train_number = 0;
int female_train_number = 0;
};
} // namespace ANN
#endif // FBC_NN_NAIVEBAYESCLASSIFIER_HPP_ naive_bayes_classifier.cpp:#include "naive_bayes_classifier.hpp"
#include "common.hpp"
#include <math.h>
#include <iostream>
#include <algorithm>
#include <fstream>
namespace ANN {
template<typename T>
int NaiveBayesClassifier<T>::init(const std::vector<sex_info<T>>& info)
{
int length = info.size();
if (length < 2) {
fprintf(stderr, "train data length should be > 1: %d\n", length);
return -1;
}
male_train_number = 0;
female_train_number = 0;
for (int i = 0; i < length; ++i) {
if (info[i].sex == 0) {
++female_train_number;
female_height.push_back(info[i].height);
female_weight.push_back(info[i].weight);
female_foot_size.push_back(info[i].foot_size);
} else {
++male_train_number;
male_height.push_back(info[i].height);
male_weight.push_back(info[i].weight);
male_foot_size.push_back(info[i].foot_size);
}
}
male_p = (T)male_train_number / (male_train_number + female_train_number);
female_p = (T)female_train_number / (male_train_number + female_train_number);
return 0;
}
template<typename T>
int NaiveBayesClassifier<T>::train(const std::string& model)
{
std::tuple<T, T> mean_variance;
calc_mean_variance(male_height, mean_variance);
male_mv.mean_height = std::get<0>(mean_variance);
male_mv.variance_height = std::get<1>(mean_variance);
calc_mean_variance(male_weight, mean_variance);
male_mv.mean_weight = std::get<0>(mean_variance);
male_mv.variance_weight = std::get<1>(mean_variance);
calc_mean_variance(male_foot_size, mean_variance);
male_mv.mean_foot_size = std::get<0>(mean_variance);
male_mv.variance_foot_size = std::get<1>(mean_variance);
calc_mean_variance(female_height, mean_variance);
female_mv.mean_height = std::get<0>(mean_variance);
female_mv.variance_height = std::get<1>(mean_variance);
calc_mean_variance(female_weight, mean_variance);
female_mv.mean_weight = std::get<0>(mean_variance);
female_mv.variance_weight = std::get<1>(mean_variance);
calc_mean_variance(female_foot_size, mean_variance);
female_mv.mean_foot_size = std::get<0>(mean_variance);
female_mv.variance_foot_size = std::get<1>(mean_variance);
CHECK(store_model(model) == 0);
return 0;
}
template<typename T>
int NaiveBayesClassifier<T>::store_model(const std::string& model) const
{
std::ofstream file;
file.open(model.c_str(), std::ios::binary);
if (!file.is_open()) {
fprintf(stderr, "open file fail: %s\n", model.c_str());
return -1;
}
file.write((char*)&male_p, sizeof(male_p));
file.write((char*)&male_mv.mean_height, sizeof(male_mv.mean_height));
file.write((char*)&male_mv.mean_weight, sizeof(male_mv.mean_weight));
file.write((char*)&male_mv.mean_foot_size, sizeof(male_mv.mean_foot_size));
file.write((char*)&male_mv.variance_height, sizeof(male_mv.variance_height));
file.write((char*)&male_mv.variance_weight, sizeof(male_mv.variance_weight));
file.write((char*)&male_mv.variance_foot_size, sizeof(male_mv.variance_foot_size));
file.write((char*)&female_p, sizeof(female_p));
file.write((char*)&female_mv.mean_height, sizeof(female_mv.mean_height));
file.write((char*)&female_mv.mean_weight, sizeof(female_mv.mean_weight));
file.write((char*)&female_mv.mean_foot_size, sizeof(female_mv.mean_foot_size));
file.write((char*)&female_mv.variance_height, sizeof(female_mv.variance_height));
file.write((char*)&female_mv.variance_weight, sizeof(female_mv.variance_weight));
file.write((char*)&female_mv.variance_foot_size, sizeof(female_mv.variance_foot_size));
file.close();
return 0;
}
template<typename T>
int NaiveBayesClassifier<T>::predict(const sex_info<T>& info) const
{
T male_height_p = calc_attribute_probability(info.height, male_mv.mean_height, male_mv.variance_height);
T male_weight_p = calc_attribute_probability(info.weight, male_mv.mean_weight, male_mv.variance_weight);
T male_foot_size_p = calc_attribute_probability(info.foot_size, male_mv.mean_foot_size, male_mv.variance_foot_size);
T female_height_p = calc_attribute_probability(info.height, female_mv.mean_height, female_mv.variance_height);
T female_weight_p = calc_attribute_probability(info.weight, female_mv.mean_weight, female_mv.variance_weight);
T female_foot_size_p = calc_attribute_probability(info.foot_size, female_mv.mean_foot_size, female_mv.variance_foot_size);
T evidence = male_p * male_height_p * male_weight_p * male_foot_size_p +
female_p * female_height_p * female_weight_p * female_foot_size_p;
T male_posterior = male_p * male_height_p * male_weight_p * male_foot_size_p /*/ evidence*/;
T female_posterior = female_p * female_height_p * female_weight_p * female_foot_size_p /*/ evidence*/;
fprintf(stdout, "male posterior probability: %e, female posterior probability: %e\n",
male_posterior, female_posterior);
if (male_posterior > female_posterior) return 1;
else return 0;
}
template<typename T>
T NaiveBayesClassifier<T>::calc_attribute_probability(T value, T mean, T variance) const
{
return (T)1 / std::sqrt(2 * PI * variance) * std::exp(-std::pow(value - mean, 2) / (2 * variance));
}
template<typename T>
int NaiveBayesClassifier<T>::load_model(const std::string& model) const
{
std::ifstream file;
file.open(model.c_str(), std::ios::binary);
if (!file.is_open()) {
fprintf(stderr, "open file fail: %s\n", model.c_str());
return -1;
}
file.read((char*)&male_p, sizeof(male_p) * 1);
file.read((char*)&male_mv.mean_height, sizeof(male_mv.mean_height) * 1);
file.read((char*)&male_mv.mean_weight, sizeof(male_mv.mean_weight) * 1);
file.read((char*)&male_mv.mean_foot_size, sizeof(male_mv.mean_foot_size) * 1);
file.read((char*)&male_mv.variance_height, sizeof(male_mv.variance_height) * 1);
file.read((char*)&male_mv.variance_weight, sizeof(male_mv.variance_weight) * 1);
file.read((char*)&male_mv.variance_foot_size, sizeof(male_mv.variance_foot_size) * 1);
file.read((char*)&female_p, sizeof(female_p)* 1);
file.read((char*)&female_mv.mean_height, sizeof(female_mv.mean_height) * 1);
file.read((char*)&female_mv.mean_weight, sizeof(female_mv.mean_weight) * 1);
file.read((char*)&female_mv.mean_foot_size, sizeof(female_mv.mean_foot_size) * 1);
file.read((char*)&female_mv.variance_height, sizeof(female_mv.variance_height) * 1);
file.read((char*)&female_mv.variance_weight, sizeof(female_mv.variance_weight) * 1);
file.read((char*)&female_mv.variance_foot_size, sizeof(female_mv.variance_foot_size) * 1);
file.close();
return 0;
}
template<typename T>
void NaiveBayesClassifier<T>::calc_mean_variance(const std::vector<T>& data, std::tuple<T, T>& mean_variance) const
{
T sum{ 0 }, sqsum{ 0 };
for (int i = 0; i < data.size(); ++i) {
sum += data[i];
}
T mean = sum / data.size();
for (int i = 0; i < data.size(); ++i) {
sqsum += std::pow(data[i] - mean, 2);
}
// unbiased sample variances
T variance = sqsum / (data.size() - 1);
std::get<0>(mean_variance) = mean;
std::get<1>(mean_variance) = variance;
}
template class NaiveBayesClassifier<float>;
template class NaiveBayesClassifier<double>;
} // namespace ANN funset.cpp:#include "funset.hpp"
#include <iostream>
#include "perceptron.hpp"
#include "BP.hpp""
#include "CNN.hpp"
#include "linear_regression.hpp"
#include "naive_bayes_classifier.hpp"
#include "common.hpp"
#include <opencv2/opencv.hpp>
// ================================ naive bayes classifier =====================
int test_naive_bayes_classifier_train()
{
std::vector<ANN::sex_info<float>> info;
info.push_back({ 6.f, 180.f, 12.f, 1 });
info.push_back({5.92f, 190.f, 11.f, 1});
info.push_back({5.58f, 170.f, 12.f, 1});
info.push_back({5.92f, 165.f, 10.f, 1});
info.push_back({ 5.f, 100.f, 6.f, 0 });
info.push_back({5.5f, 150.f, 8.f, 0});
info.push_back({5.42f, 130.f, 7.f, 0});
info.push_back({5.75f, 150.f, 9.f, 0});
ANN::NaiveBayesClassifier<float> naive_bayes;
int ret = naive_bayes.init(info);
if (ret != 0) {
fprintf(stderr, "naive bayes classifier init fail: %d\n", ret);
return -1;
}
const std::string model{ "E:/GitCode/NN_Test/data/naive_bayes_classifier.model" };
ret = naive_bayes.train(model);
if (ret != 0) {
fprintf(stderr, "naive bayes classifier train fail: %d\n", ret);
return -1;
}
return 0;
}
int test_naive_bayes_classifier_predict()
{
ANN::sex_info<float> info = { 6.0f, 130.f, 8.f, -1 };
ANN::NaiveBayesClassifier<float> naive_bayes;
const std::string model{ "E:/GitCode/NN_Test/data/naive_bayes_classifier.model" };
int ret = naive_bayes.load_model(model);
if (ret != 0) {
fprintf(stderr, "load naive bayes classifier model fail: %d\n", ret);
return -1;
}
ret = naive_bayes.predict(info);
if (ret == 0) fprintf(stdout, "It is a female\n");
else fprintf(stdout, "It is a male\n");
return 0;
} 执行结果如下:

GitHub: https://github.com/fengbingchun/NN_Test
相关文章推荐
- 吴恩达老师深度学习视频课笔记:单隐含层神经网络公式推导及C++实现(二分类)
- 拉格朗日插值算法简介及其C++实现
- C++队列实现和队列简介(不是循环队列)
- 栈的简介与C++模板实现
- 队列的简介与C++模板实现
- Levenberg-Marquardt算法简介和C++实现
- 【机器学习实战之二】:C++实现基于概率论的分类方法--朴素贝叶斯分类(Naive Bayes Classifier)
- Hypertable 简介 一个 C++ 的Bigtable开源实现
- 【C++】模版实现双向链表的各种操作(如:逆置、去重Unique、分类(冒泡)、合并)
- gabor 滤波的c++实现与该类得使用简介
- 朴素的矩阵行列式计算之C++实现
- 决策树C4.5分类算法的C++实现
- C++实现分类统计字符串中字符个数
- 卡尔曼滤波简介及其算法实现代码(C++/C/MATLAB)
- 工厂模式简介(C++实现)
- 转 -- Hypertable 简介 (一个 C++ 的Bigtable开源实现)
- 卡尔曼滤波简介及其算法实现代码(C++/C/MATLAB)
- 朴素贝叶斯分类器原理与java实现
- Hypertable 简介 (一个 C++ 的Bigtable开源实现) 【很好的入门的例子】
- 单例模式简介以及C++版本的实现
