Oncomine: 一个肿瘤相关基因研究的数据库--转载
2017-02-13 13:48
411 查看
如果你获得了一个肿瘤差异表达基因,想研究其是否可作为某种肿瘤的潜在标志物和靶点,又怕做实验会得到阴性结果,浪费时间和金钱,这时候你就应该想到Oncomine数据库了(www.oncomine.org)。
Oncomine整合了GEO、TCGA和已发表的文献等来源的RNA和DNA-seq数据,只要你用非营利机构邮箱注册了就可以免费使用了。下面就以ERBB2基因为例跟着小诺一步一步搞定Oncomine吧。
第一步:ERBB2在肿瘤中的表达
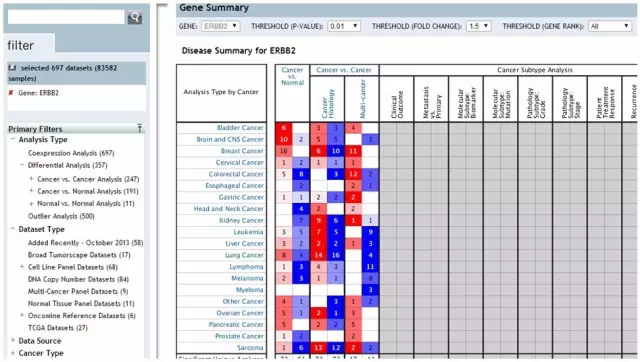
在搜索框中输入ERBB2并设定P值、差异表达倍数和差异的排序就可以得到这个基因在各类肿瘤中的差异表达数据。如下图所示,在有差异表达的数据中,ERBB2在膀胱癌、脑和神经瘤、乳腺癌和前列腺癌中高表达的几率较高(红色表示高表达,颜色越深表达量越高,蓝色反之)。
第二步:ERBB2在乳腺癌中的表达
为进一步分析ERBB2在我感兴趣的乳腺癌中的表达,点击左侧的数据筛选区里选择Breast cancer、Cancer vs. Normal和Clinical Specimen即可知道其在乳腺癌组织中的表达量是否升高。

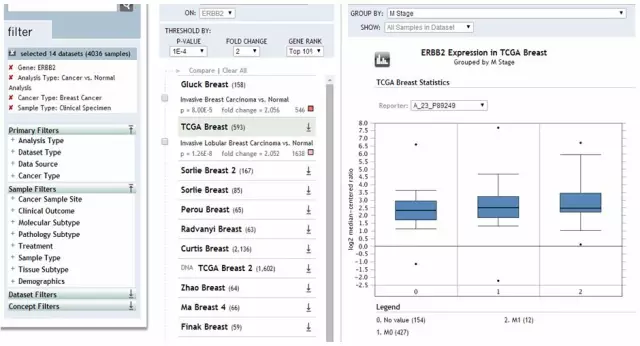
第三步:ERBB2的表达与乳腺癌TNM分期、分化和生存时间等临床病理特点及预后的关系
点击样本量较大、数据可信度高的TCGA Breast或Curtis Breast,在GROUPED BY窗口中选择相应的临床资料分组就可得出ERBB2在相应的临床资料分组中的表达量。
第四步:ERBB2共表达基因分析
在Analysis Type中选择Coexpression Analysis,即可获得与ERBB2表达正相关性较高的基因。

第五步:寻找差异表达基因
若是还不知道从哪个基因下手,没关系,Oncomine也能帮你解决。在左侧选择Cancer Type和Cancer vs. Normal即可得到差异表达基因。

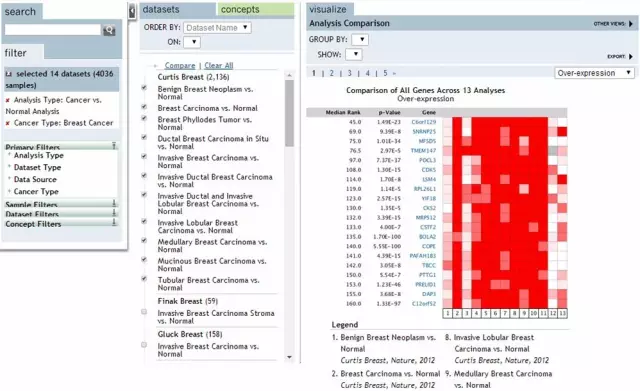
选择多个乳腺癌,点击compare即可获得多个数据中肿瘤高表达基因。
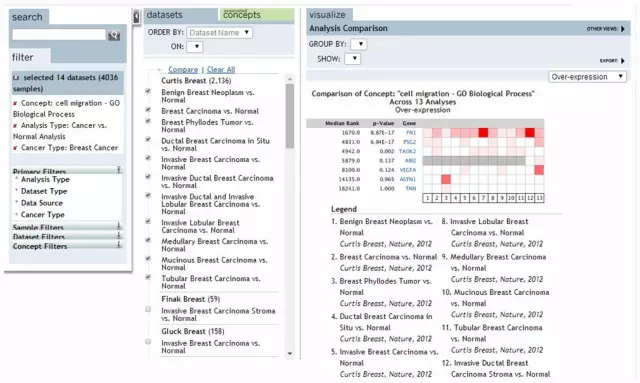
另外在搜索框中输入如Kinase、TGFbeta signaling或是cell migration即可获得相应的激酶、信号通路或是Go富集分析相关高表达基因。
以上就是Oncomine在肿瘤研究中的基本功能,当然还可以获取其它有用的信息如药物敏感相关基因,我这里就不一一介绍了。大家可以结合自己的研究方向和兴趣进行搜索和筛选。
转载本文请联系原作者获取授权,同时请注明本文来自熊朝亮科学网博客。
链接地址:http://blog.sciencenet.cn/blog-1509670-986593.html
Oncomine整合了GEO、TCGA和已发表的文献等来源的RNA和DNA-seq数据,只要你用非营利机构邮箱注册了就可以免费使用了。下面就以ERBB2基因为例跟着小诺一步一步搞定Oncomine吧。
第一步:ERBB2在肿瘤中的表达
在搜索框中输入ERBB2并设定P值、差异表达倍数和差异的排序就可以得到这个基因在各类肿瘤中的差异表达数据。如下图所示,在有差异表达的数据中,ERBB2在膀胱癌、脑和神经瘤、乳腺癌和前列腺癌中高表达的几率较高(红色表示高表达,颜色越深表达量越高,蓝色反之)。

第二步:ERBB2在乳腺癌中的表达
为进一步分析ERBB2在我感兴趣的乳腺癌中的表达,点击左侧的数据筛选区里选择Breast cancer、Cancer vs. Normal和Clinical Specimen即可知道其在乳腺癌组织中的表达量是否升高。

第三步:ERBB2的表达与乳腺癌TNM分期、分化和生存时间等临床病理特点及预后的关系
点击样本量较大、数据可信度高的TCGA Breast或Curtis Breast,在GROUPED BY窗口中选择相应的临床资料分组就可得出ERBB2在相应的临床资料分组中的表达量。

第四步:ERBB2共表达基因分析
在Analysis Type中选择Coexpression Analysis,即可获得与ERBB2表达正相关性较高的基因。

第五步:寻找差异表达基因
若是还不知道从哪个基因下手,没关系,Oncomine也能帮你解决。在左侧选择Cancer Type和Cancer vs. Normal即可得到差异表达基因。

选择多个乳腺癌,点击compare即可获得多个数据中肿瘤高表达基因。

另外在搜索框中输入如Kinase、TGFbeta signaling或是cell migration即可获得相应的激酶、信号通路或是Go富集分析相关高表达基因。

以上就是Oncomine在肿瘤研究中的基本功能,当然还可以获取其它有用的信息如药物敏感相关基因,我这里就不一一介绍了。大家可以结合自己的研究方向和兴趣进行搜索和筛选。
转载本文请联系原作者获取授权,同时请注明本文来自熊朝亮科学网博客。
链接地址:http://blog.sciencenet.cn/blog-1509670-986593.html
相关文章推荐
- 【转载】通过SQL获取MSSQL的数据库相关信息收藏
- 转:把一个java web应用包装成桌面应用的简单做法(个人比较喜欢,想有空研究一下,特意转载,以备后用)
- 数据库相关文章转载(2) MySQL自带的性能压力测试工具mysqlslap详解
- 云数据库研究 相关论文阅读
- (转载)与OpenDialog相关的一个问题
- 一个程序详细研究DataReader (转载)
- 【转载苏飞的博客】一个操作数据库的类(备份,还原,分离,附加,添加删除用户等操作)
- 转载:数据库的相关书籍
- FusionCancer-人类癌症相关的融合基因的数据库
- 染色体、基因与人体基因组研究(转载于新华网)
- IPhone短信相关部分研究(转载)
- 最近在写研究jmemcache,想实现一个数据库和数据库请求的中间程序
- 数据库相关文章转载(1) MySQL性能优化之参数配置
- 转载 :一个Job运行失败导致数据库挂死
- C#多线程学习(二) 如何操纵一个线程 (转载系列)——继续搜索引擎研究
- C#多线程学习(一) 多线程的相关概念(转载系列)——继续搜索引擎研究
- [置顶] 转载一个即将成为大牛朋友的学习方法(内涵相关文档资料,java相关),
- 转载一个别人收藏的精典网站Ruby,HIBERNATE相关
- 简单粗暴,微生物生态研究中常用数据库简介--转载
- 五花八门的信用与业务相关程度研究是一个思路
